Structure of the mycobacterial ESX-5 type VII secretion system pore complex
Science advances 25 June 2021
10.1126/sciadv.abg9923
EMBL Hamburg’s Wilmanns and Kosinski groups have determined the detailed structure of a bacterial protein complex critical for tuberculosis infection
Tuberculosis is one of the top ten causes of death worldwide, infecting about one-quarter of the world’s population. Although it is treatable, the rise of multidrug-resistant tuberculosis poses a major threat to global health security, and has been declared by the World Health Organization as a global health emergency. Reduced access to diagnosis and treatment during the COVID-19 pandemic is expected to dramatically increase the number of tuberculosis infections. This will set global efforts to tackle the disease back several years.
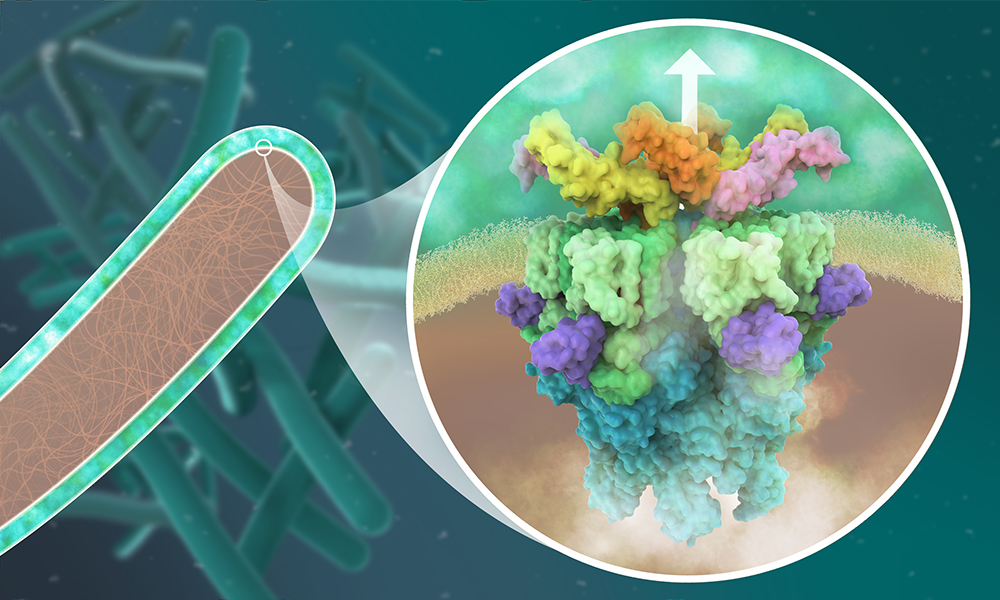
Tuberculosis is caused by infection with Mycobacterium tuberculosis: a bacterium that infects human lungs and other organs by using complex molecular machineries. These include protein complexes known as type VII secretion systems, which enable M. tuberculosis to release molecules into its host, which disarm and ultimately kill the infected human cell. Five such secretion systems, labelled ESX-1 to ESX-5, are found among M. tuberculosis and other closely related mycobacteria, many of which are pathogenic. Without them, the bacteria are unable to infect human cells.
The Wilmanns group at EMBL Hamburg has been using high-resolution structural biology to study mycobacterial proteins for the last two decades. The molecular understanding of the bacterial machinery used to infect cells resulted in collaborations with industry to develop new drugs against tuberculosis. In their most recent study, they determined the molecular structure of the secretion system ESX-5 to a high level of detail. They saw that the core of ESX-5 is built of 30 protein units, which form a dynamic membrane pore to allow secretion of proteins that enable the bacterium to survive and multiply inside human cells. Knowledge of the ESX-5 structure at high-resolution is essential to target specific sites with small-molecule drugs.
“Our new structure of the ESX-5 secretion complex provides deep insight into a major sluice gate that separates the inner of these bacteria from the outer host environment. Opening this gate allow the pathogen to spit out its deadly weapons to infect humans to develop tuberculosis. We can use this structure as a toolbox with literally thousands of potential drug targets. This will open an entirely new field of studies on tuberculosis,” says Matthias Wilmanns, who leads the study. Kate Beckham, who developed an innovative way to isolate ESX-5, adds: “The central pore we saw in ESX-5 could serve as a new drug target. Blocking it could prevent infection with pathogenic mycobacteria.”
The study could also help scientists to develop new vaccines for tuberculosis. The widely used Bacillus Calmette–Guérin (BCG) vaccine, which has its 100th anniversary this year, is based on a strain of mycobacterium that has lost its ability to cause disease because of a defect in the ESX-1 system. However, as BCG vaccination offers insufficient protection and is most effective in young infants only, alternative vaccines are urgently needed. Due to its close structural and functional relation with ESX-1, targeting the ESX-5 secretion system might spur the development of new vaccines that could complement or replace those currently used.
Determining the molecular structure of ESX-5 was particularly challenging because of its large size and complexity. No single structural biology method can provide the full picture. In this case, the key to success was using integrative structural biology, in which data obtained using different methods – cryo-electron microscopy, X-ray crystallography, mass spectrometry and computational methods – were used jointly to create a coherent model.
“Eighteen months ago, solving this structure looked like mission: impossible,” says Matthias Wilmanns. “We managed to put the puzzle pieces together because each team member contributed unique expertise. To solve the complete structure, we collaborated with Jan Kosinski’s group at EMBL Hamburg and the Centre for Structural Systems Biology, which provided necessary expertise in integrative structural biology. We also received great help from our colleagues at EMBL Heidelberg, who performed cryo-electron microscopy experiments.”
This study illustrates some of EMBL’s approaches to life science research in its forthcoming scientific programme, Molecules to Ecosystems 2022–2026. As part of this programme, EMBL will take an interdisciplinary approach to understanding the molecular basis of life in the context of environmental changes. This will provide translational potential to support advances in human and planetary health.
EMBL’s approach, including this study, is aligned with the collaborative efforts of other research groups and institutions from Northern Germany working together at the Centre for Structural Systems Biology.

Obwohl sie in den meisten Fällen gut behandelbar ist, ist Tuberkulose eine der zehn häufigsten Todesursachen weltweit. Ein Viertel der Weltbevölkerung ist mit Tuberkulose infiziert. Die Verbreitung der multiresistenten Tuberkulose stellt eine große Bedrohung für die globale Gesundheitssicherheit dar und wurde von der Weltgesundheitsorganisation zum globalen Gesundheitsnotstand erklärt. Der zusätzlich eingeschränkte Zugang zu Diagnose und Behandlung während der COVID-19-Pandemie erhöht die Zahl der Tuberkulose-Infektionen voraussichtlich dramatisch und wird damit globale Bemühungen zur Bekämpfung der Krankheit um mehrere Jahre zurückwerfen.
Tuberkulose wird durch eine Infektion mit dem Mycobacterium tuberculosis verursacht: einem Bakterium, das die menschliche Lunge und andere Organe mit Hilfe komplexer molekularer Mechanismen infiziert. Dazu gehören Proteinkomplexe, die als Typ-VII-Sekretionssysteme bekannt sind und die es M. tuberculosis ermöglichen, Moleküle in seinem Wirt freizusetzen, die die infizierte menschliche Zelle entwaffnen und schließlich abtöten. Fünf solcher Sekretionssysteme, bezeichnet als ESX-1 bis ESX-5, finden sich bei M. tuberculosis und anderen eng verwandten, meist pathogenen Mykobakterien. Ohne die Sekretionssysteme sind die Bakterien nicht in der Lage, menschliche Zellen zu infizieren.
Die Wilmanns Gruppe am EMBL Hamburg hat in den vergangenen zwei Jahrzehnten mit Hilfe der hochauflösenden Methoden der Strukturbiologie mykobakterielle Proteine untersucht. In ihrer jüngsten Studie bestimmte sie die molekulare Struktur des Sekretionssystems ESX-5 im kleinstmöglichen Detail. Die ForscherInnen fanden dabei heraus, dass der Kern von ESX-5 aus 30 Proteineinheiten aufgebaut ist, die eine dynamische Membranpore bilden um die Sekretion von Proteinen zu ermöglichen, die dem Bakterium das Überleben und die Vermehrung in menschlichen Zellen ermöglichen. Die Kenntnis der ESX-5-Struktur in hoher Auflösung ist unerlässlich, um spezifische Stellen mit niedermolekularen Wirkstoffen anzusprechen. Das molekulare Verständnis der bakteriellen Mechanismen, mit der Zellen infiziert werden, führte bereits zu Kooperationen mit der Industrieforschung, um neue Medikamente gegen Tuberkulose zu entwickeln.
“Unsere neue Struktur des ESX-5-Sekretionskomplexes gibt einen tiefen Einblick in ein wichtiges Schleusentor, das das Innere dieser Bakterien von der äußeren Wirtsumgebung trennt. Wird dieses Tor geöffnet, kann der Erreger seine tödlichen Waffen ausspucken, um den Menschen zu infizieren und Tuberkulose zu entwickeln. Wir können diese Struktur als Werkzeugkasten mit buchstäblich Tausenden von potenziellen Wirkstoffzielen nutzen. Damit eröffnet sich ein völlig neues Feld für Studien zur Tuberkulose”, sagt Matthias Wilmanns, der die Studie leitet. Kate Beckham, die einen innovativen Weg zur Isolierung von ESX-5 entwickelt hat, ergänzt: “Die zentrale Pore, die wir in ESX-5 gesehen haben, könnte als neues Ziel für Medikamente dienen. Sie zu blockieren könnte eine Infektion mit pathogenen Mykobakterien verhindern.”
Die Studie könnte Wissenschaftlern auch dabei helfen, neue Impfstoffe gegen Tuberkulose zu entwickeln. Der weit verbreitete Bacillus Calmette-Guérin (BCG)-Impfstoff, der in diesem Jahr sein 100-jähriges Jubiläum feiert, basiert auf einem Mykobakterienstamm, der durch einen Defekt im ESX-1-System seine Fähigkeit verloren hat, Krankheiten zu verursachen. Da die BCG-Impfung mittlerweile also nur einen unzureichenden Schutz bietet und lediglich bei Säuglingen am wirksamsten ist, werden dringend alternative Impfstoffe benötigt. Aufgrund seiner engen strukturellen und funktionellen Verwandtschaft mit ESX-1 könnte die gezielte Beeinflussung des ESX-5-Sekretionssystems die Entwicklung neuer Impfstoffe vorantreiben, die die derzeit verwendeten Impfstoffe ergänzen oder ersetzen könnten.
Die Bestimmung der molekularen Struktur von ESX-5 war aufgrund seiner großen Größe und Komplexität eine besondere Herausforderung. Keine einzelne strukturbiologische Methode kann das vollständige Bild liefern. In diesem Fall lag der Schlüssel zum Erfolg in der integrativen Strukturbiologie, bei der Daten, die mit verschiedenen Methoden gewonnen wurden – Kryo-Elektronenmikroskopie, Röntgenkristallographie, Massenspektrometrie und Berechnungsmethoden – übereinander gelegt wurden, um ein kohärentes Modell zu erstellen. Der in der vorliegenden Studie angewandte Arbeitsansatz ist auf die Zusammenarbeit mit verschiedenen Forschungsgruppen und Institutionen abgestimmt, die im Hamburger Zentrum für Strukturelle Systembiologie (CSSB) zusammenarbeiten.
“Vor achtzehn Monaten sah die Lösung dieser Struktur wie eine ‚Mission: impossible‘ aus”, sagt Matthias Wilmanns. “Wir haben es geschafft, die Puzzlestücke zusammenzusetzen, weil jedes Teammitglied einzigartige Expertise beisteuerte. Um die komplette Struktur zu lösen, arbeiteten wir mit Jan Kosinskis Gruppe am EMBL Hamburg und dem Zentrum für Strukturelle Systembiologie zusammen, die die notwendige Expertise in integrativer Strukturbiologie beisteuerten. Große Hilfe erhielten wir auch von unseren Kollegen am EMBL Heidelberg, die kryo-elektronenmikroskopische Experimente durchführten.”
Diese Studie veranschaulicht einige der Ansätze, die das EMBL im Rahmen seines kommenden wissenschaftlichen Programms “Molecules to Ecosystems 2022-2026” in der lebenswissenschaftlichen Forschung verfolgt. Im Rahmen dieses Programms wird das EMBL einen interdisziplinären Ansatz verfolgen, um die molekularen Grundlagen des Lebens im Kontext von Umweltveränderungen zu verstehen. Dies wird das translationales Potenzial bieten, Fortschritte in der globalen Gesundheits- und Umweltforschung zu unterstützen.
Science advances 25 June 2021
10.1126/sciadv.abg9923
Looking for past print editions of EMBLetc.? Browse our archive, going back 20 years.
EMBLetc. archive