Dynamic assembly of a large multidomain ribozyme visualized by cryo-electron microscopy
Nature Communications 11November 2025
https://doi.org/10.1038/s41467-025-65502-8
Researchers have visualised, in unprecedented detail, how a large RNA molecule assembles itself into a functional machine
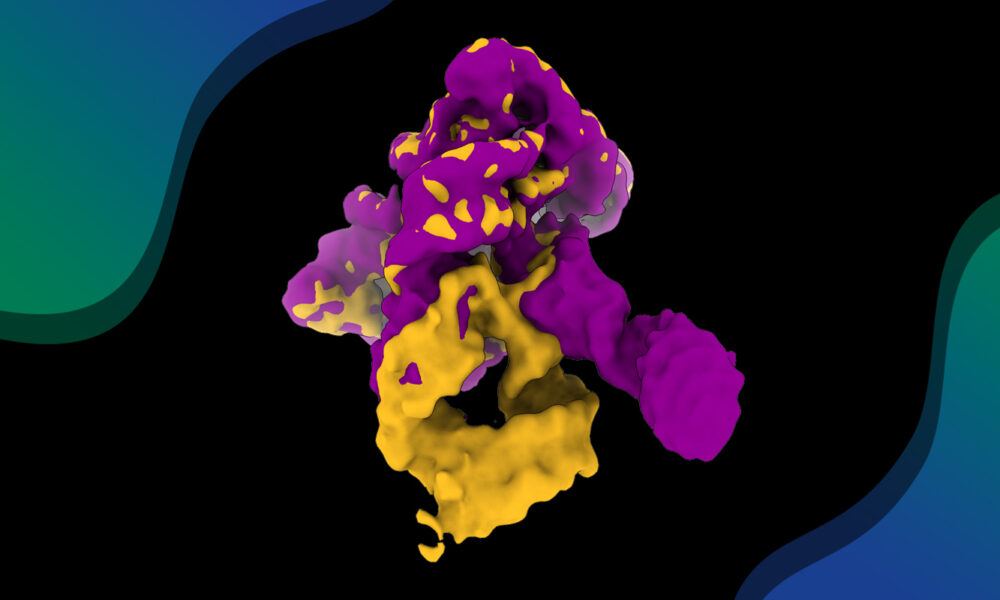
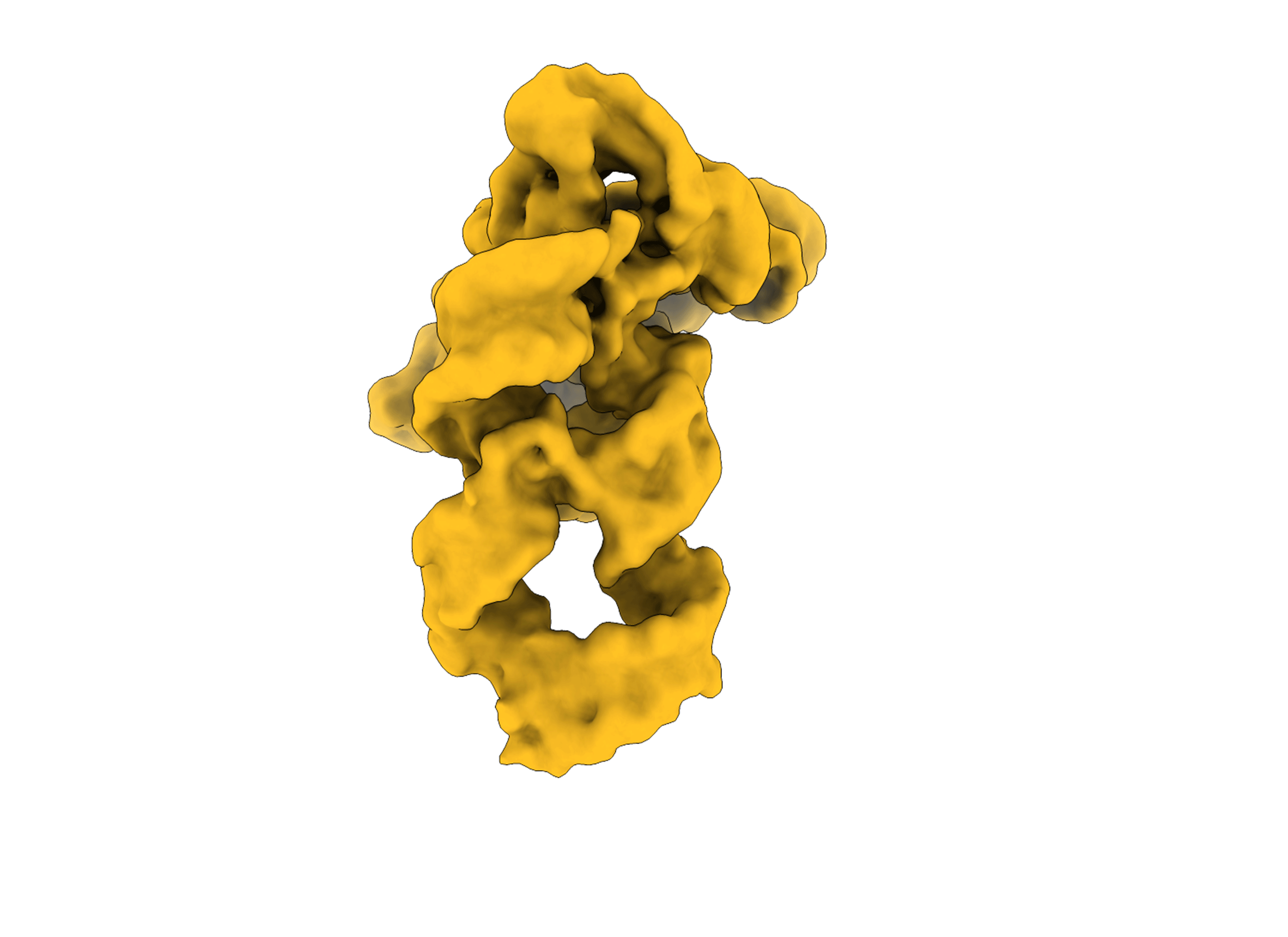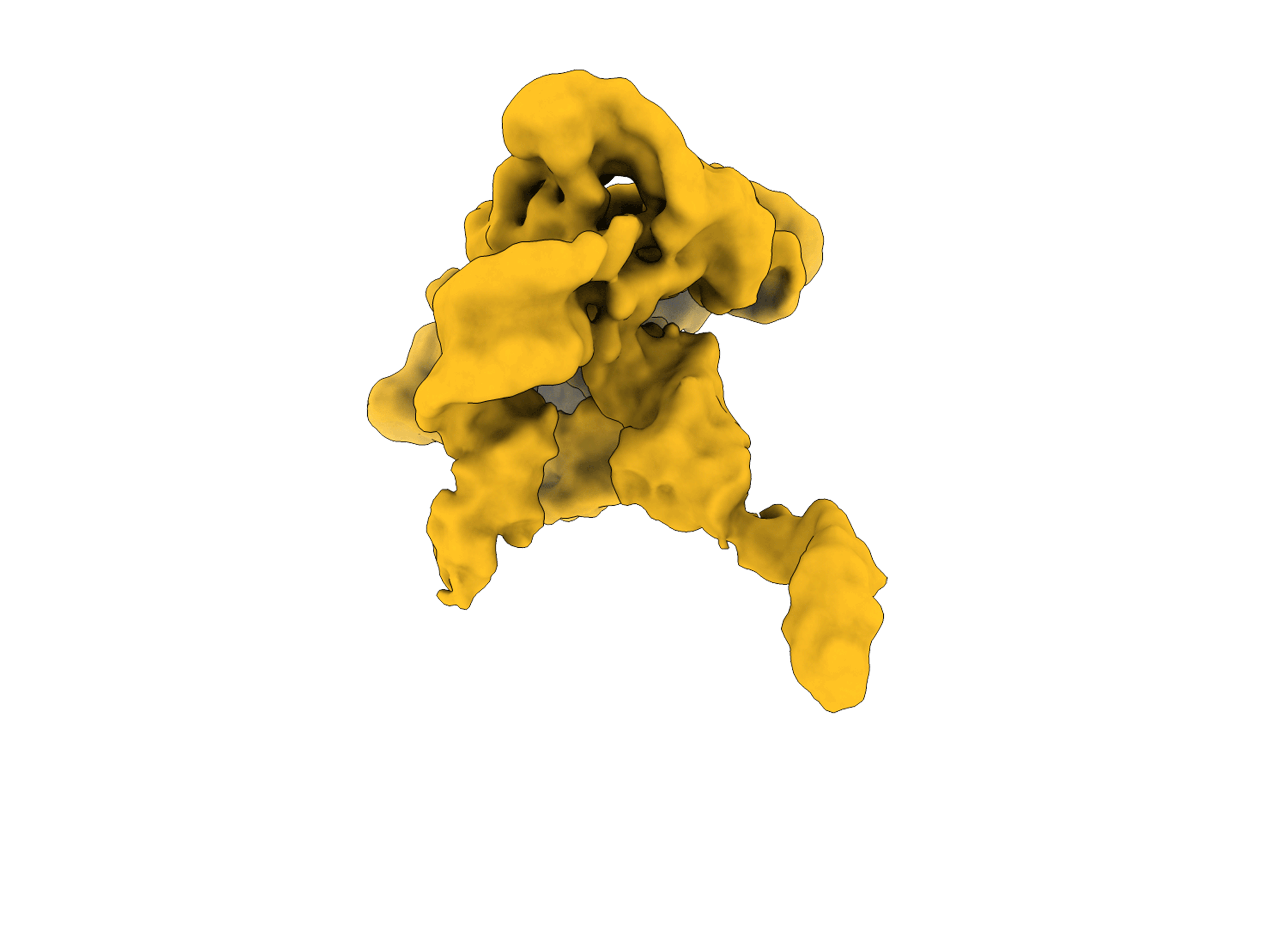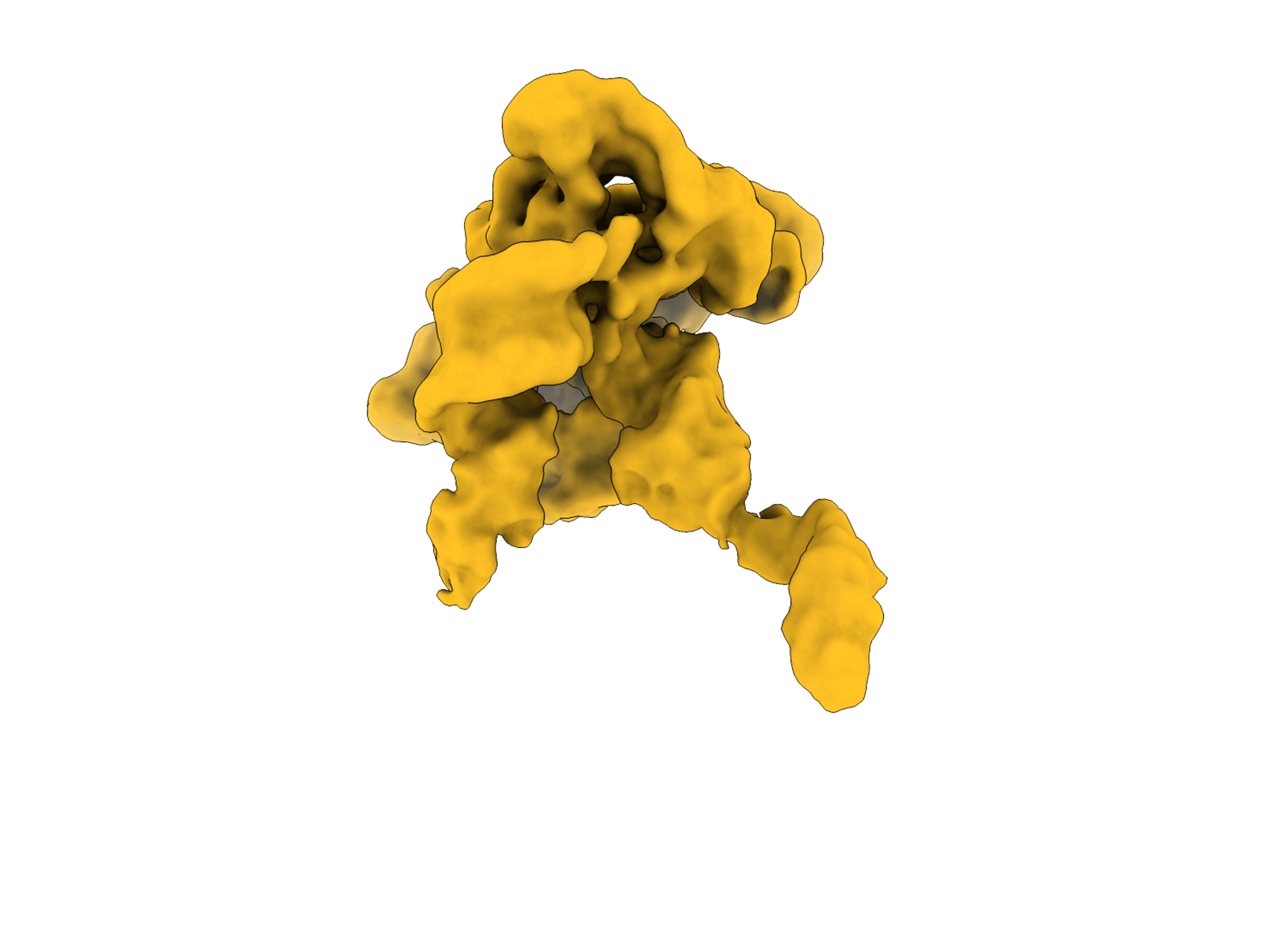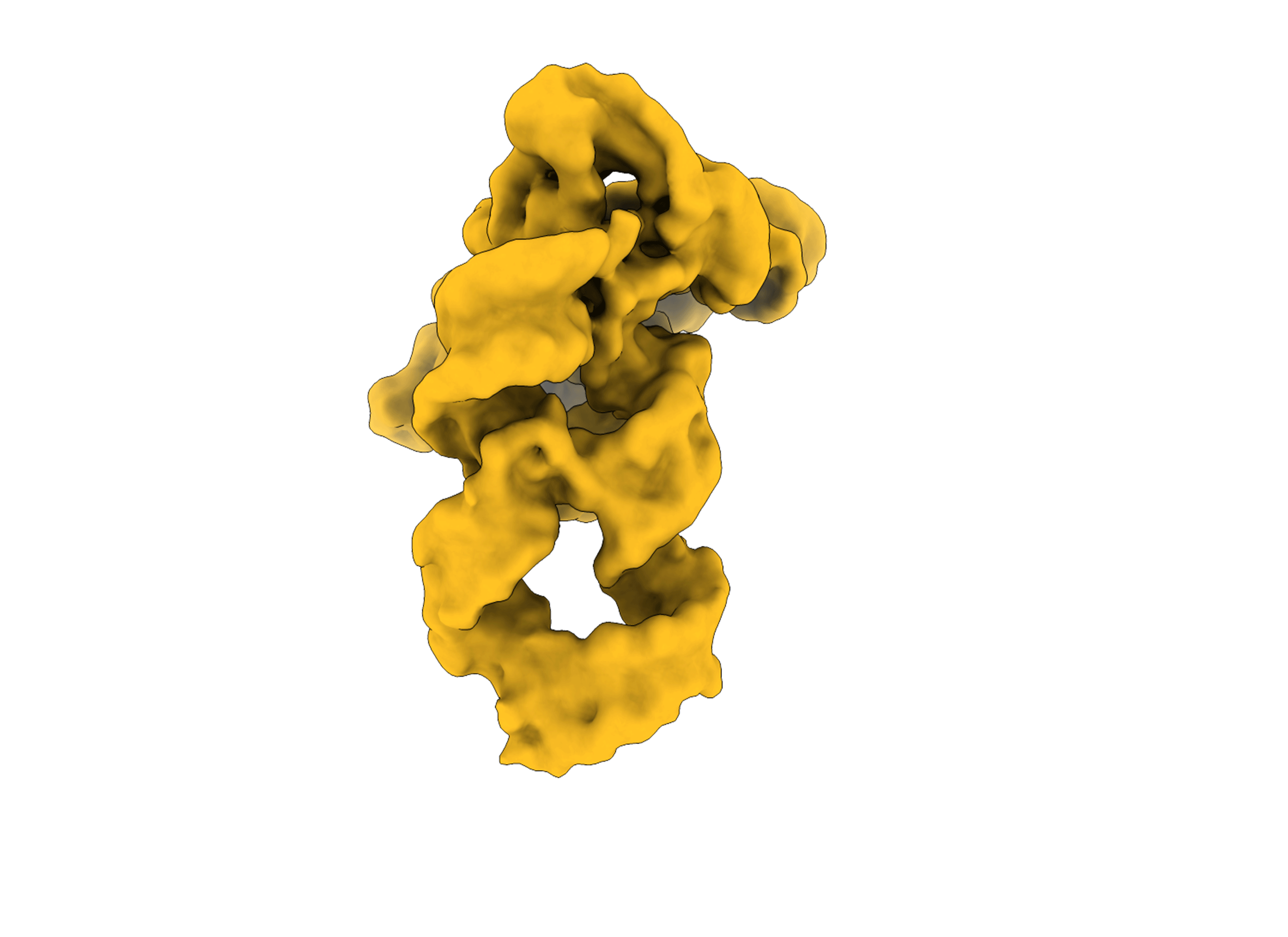
RNA is a central biological molecule, now widely harnessed in medicine and nanotechnology. Like proteins, RNA often gets its function from its three-dimensional structure. A recent study in Nature Communications has captured, for the first time, a ribozyme in motion — almost frame by frame. The researchers recorded how this tiny RNA machine folds, flexes, and assembles itself, revealing its intricate choreography in unprecedented detail.
Using state-of-the-art techniques — cryo-electron microscopy (cryo-EM), small-angle X-ray scattering (SAXS), RNA biochemistry and enzymology, image processing, and molecular simulations — the scientists observed the assembly of a self-splicing ribozyme – an RNA molecule that can ‘cut and paste’ its own sequence, essentially editing itself to become operational. They captured the dynamic ‘behind-the-scenes’ process by which the self-splicing ribozyme folds into its functional structure. The research was led by the team of Marco Marcia, former EMBL Group Leader and currently Associate Professor and SciLifeLab Group leader at Uppsala University, Sweden.
This breakthrough was made possible thanks to the cutting-edge facilities and expert services at EMBL Grenoble, which enabled the integration of advanced structural biology methods with RNA biochemistry and enzymology. The Marcia group also benefited from close collaboration with the Centre for Structural Systems Biology (CSSB) Hamburg, where innovative cryo-EM image processing approaches tailored for this specific project were developed, and the Istituto Italiano di Tecnologia (IIT), which provided high-level molecular simulation expertise.
“Determining RNA structures is a challenging task – the inherent flexibility and negative charge make RNA a notoriously difficult target for structural studies,” said Shekhar Jadhav, former Predoctoral Fellow at EMBL Grenoble, now a postdoc at Uppsala University, Sweden. “Persistent efforts and extensive screening on electron microscopes ultimately led us to visualise elusive RNA dynamics.”
At the heart of this production is Domain 1 (D1), the ribozyme’s central scaffold and, as it turns out, its director. This domain acts as a molecular gate, cueing the other domains (D2, D3, D4) to enter at precisely the right moment during the folding process.
Subtle movements in key parts of the D1 molecule prompt one of its sections to open up and make way for the next. Each domain joins the scene only when the previous one is correctly in place, creating a seamless sequence of molecular choreography that prevents structural errors and ensures a flawless finale: the formation of a structure that can catalyse a chemical reaction, essential to the ribozyme’s function.
By analysing hundreds of thousands of single RNA particles, the team reconstructed intermediate ‘takes’ that were invisible in static crystal structures. These fleeting frames show how the RNA explores alternative poses before settling into its final conformation.
“To capture these fleeting frames, we had to develop novel cryo-EM image-processing strategies,” said Maya Topf, Group Leader at CSSB, Professor at the University Medical Centre Hamburg-Eppendorf, and a collaborator on the study. “This is a great example of how computational innovation and high-quality cryoEM data can reveal the hidden conformations of molecular machines.”
SAXS data and molecular dynamics simulations helped the scientists refine each frame and assemble the storyline.
“One major strength of this work is the synergy between these cutting-edge new structural data on RNA and our advanced molecular simulations of this challenging system,” said Marco De Vivo, Head of Molecular Modeling and Drug Discovery Lab and Associate Director for Computation of Institu Italiano di Technologia in Genoa, and one of the collaborators on this study. “This combined approach has clarified, at an unprecedented atomistic level of detail, the dynamic that drives the entire assembly of this RNA molecule, which now opens new avenues for drug discovery efforts targeting RNA.”
Group II introns, the ribozymes featured in this molecular film, are thought to be the ancestors of the spliceosome, the complex machinery that edits RNA in human cells. By revealing how these molecules fold efficiently and avoid kinetic traps, the study provides new insight into how early RNA-based life may have evolved its editing tools. Beyond evolutionary lore, this work also sets the stage for RNA design and engineering – guiding how future biotechnologies might script RNA molecules to fold correctly for use in therapeutics or nanobiotechnology.
The detailed datasets and molecular mechanisms uncovered in this study offer a valuable benchmark for training and testing AI models. Some of the RNA structures resolved here have already been used in international CASP competitions — the same predictive challenge that gave rise to AlphaFold — as recently described in the journal Proteins.
“This work is expected to play a key role in shaping artificial intelligence approaches to RNA structure prediction, paving the way towards a new ‘AlphaFold for RNA’.” said Marcia.
This convergence of experimental precision and machine learning marks a new phase for RNA structural biology, where AI and cryo-EM can learn from each other to predict, visualise, and understand the dynamics of life’s most versatile molecule.
L’ARN est une molécule biologique essentielle, aujourd’hui largement exploitée en médecine et en nanotechnologie. À l’instar des protéines, l’ARN tire souvent sa fonction de sa structure tridimensionnelle. Une étude récente, publiée dans Nature Communications, a pour la première fois capturé un ribozyme en mouvement — presque image par image. Les chercheurs ont enregistré la façon dont cette minuscule machine d’ARN se plie, se déplie et s’assemble, révélant une chorégraphie d’une complexité inédite et d’un niveau de détail sans précédent.
En utilisant des techniques de pointe — cryo-microscopie électronique (cryo-EM), diffusion des rayons X aux petits angles (SAXS), biochimie et enzymologie de l’ARN, traitement d’images et simulations moléculaires — les scientifiques ont observé l’assemblage d’un ribozyme auto-épisseur, une molécule d’ARN capable de « couper et coller » sa propre séquence, pour s’éditer en quelque sorte pour devenir opérationnelle. Ils ont capturé le processus dynamique et « en coulisses » par lequel le ribozyme auto-épisseur se replie pour adopter sa structure fonctionnelle. La recherche a été menée par l’équipe de Marco Marcia, ancien chef d’équipe à l’EMBL et actuellement professeur associé et chef de groupe SciLifeLab à l’Université d’Uppsala, en Suède.
Cette avancée a été rendue possible grâce aux infrastructures de pointe et à l’expertise des services de l’EMBL Grenoble, qui ont permis d’intégrer des approches avancées de biologie structurale à la biochimie et à l’enzymologie de l’ARN. L’équipe Marcia a également bénéficié d’une collaboration étroite avec le Center for Structural Systems Biology (CSSB) à Hambourg, où des méthodes innovantes de traitement d’images en cryo-EM ont été développées spécifiquement pour ce projet, ainsi qu’avec l’Istituto Italiano di Tecnologia (IIT), qui a apporté son expertise en simulations moléculaires.
« Déterminer la structure de l’ARN est une tâche ardue : sa flexibilité intrinsèque et sa charge négative en font une cible difficile pour les études structurales », explique Shekhar Jadhav, ancien doctorant à l’EMBL Grenoble et aujourd’hui postdoctorant à l’Université d’Uppsala, en Suède. « Des efforts soutenus et un criblage approfondi au microscope électronique nous ont finalement permis de visualiser des dynamiques de l’ARN jusque-là insaisissables. »
De cela en résulte le “film moléculaire” le plus complet à ce jour montrant une molécule d’ARN en train de se construire elle-même, révélant comment elle évite l’équivalent biologique des prises ratées : des états mal repliés et non fonctionnels, connus sous le nom de pièges cinétiques.
Au cœur de cette chorégraphie se trouve le Domaine 1 (D1), le support central du ribozyme et, en quelque sorte, son metteur en scène. Ce domaine agit comme une entrée de scène moléculaire, donnant le signal aux autres domaines (D2, D3, D4) d’entrer à un moment exact lors du repliement.
De subtils mouvements au sein de régions clés de la molécule D1 incitent l’une de ses sections à s’ouvrir pour laisser passer la suivante. Chaque domaine n’entre en scène que lorsque le précédent est correctement positionné, créant une séquence fluide de chorégraphie moléculaire qui prévient les erreurs structurelles et garantit un final sans faute : la formation d’une structure capable de catalyser une réaction chimique, indispensable à la fonction du ribozyme.
En combinant plusieurs méthodes d’analyse sur des centaines de milliers de particules individuelles, l’équipe a pu reconstruire des « prises » intermédiaires invisibles dans les structures cristallines, statiques usuellement observées par cristallographie à rayon X. Ces images fugaces montrent comment l’ARN explore des positions alternatives avant de se stabiliser dans sa conformation finale.

« Pour capturer ces mouvements fugaces, nous avons dû développer de nouvelles stratégies de traitement d’images en cryo-EM », explique Maya Topf, cheffe d’équipe au CSSB, professeure au Centre médical universitaire de Hambourg-Eppendorf et collaboratrice de l’étude. « C’est un excellent exemple de la manière dont l’innovation computationnelle et des données cryo-EM de haute qualité peuvent révéler les conformations cachées des machines moléculaires. »
Les données de SAXS et les simulations de dynamique moléculaire ont aidé les scientifiques à affiner chaque « image » et à assembler le fil narratif.
« L’un des grands atouts de ce travail réside dans la synergie entre ces nouvelles données structurales de pointe sur l’ARN et nos simulations moléculaires avancées de ce système complexe », souligne Marco De Vivo, responsable du laboratoire de modélisation moléculaire et découverte de médicaments, et directeur associé pour le calcul à l’Institut Italien de Technologie de Gênes, ainsi que l’un des collaborateurs de l’étude. « Cette approche combinée a permis de clarifier, avec un niveau de détail atomistique jamais atteint, la dynamique qui gouverne l’assemblage complet de cette molécule d’ARN — ouvrant ainsi de nouvelles perspectives pour la découverte de médicaments ciblant l’ARN. »
Les introns de groupe II, les ribozymes mis en scène dans ce film moléculaire, sont considérés comme les ancêtres du spliceosome, la machinerie complexe qui édite l’ARN dans les cellules humaines. En montrant comment ces molécules se replient efficacement et évitent les pièges cinétiques, l’étude offre un nouvel éclairage sur la manière dont les premières formes de vie fondées sur l’ARN ont pu développer leurs outils d’édition.
Au-delà de l’aspect évolutif, ces travaux préparent également le terrain pour la conception et l’ingénierie de l’ARN : ils orientent la manière dont les biotechnologies de demain pourront programmer des molécules d’ARN capables de se replier correctement pour des usages thérapeutiques ou en nanobiotechnologie.
Les jeux de données détaillés et les mécanismes moléculaires mis au jour dans cette étude constituent une référence précieuse pour l’entraînement et l’évaluation des modèles d’IA. Certaines des structures d’ARN résolues ici ont déjà été utilisées dans les compétitions internationales CASP — le même défi prédictif qui a donné naissance à AlphaFold — comme récemment décrit dans la revue Proteins.
« Ces travaux devraient jouer un rôle clé dans l’élaboration d’approches d’intelligence artificielle dédiées à la prédiction de la structure de l’ARN, ouvrant la voie vers un nouvel “AlphaFold de l’ARN” », déclare Marcia.
Cette convergence entre précision expérimentale et apprentissage automatique marque une nouvelle étape pour la biologie structurale de l’ARN, où l’IA et la cryo-EM peuvent s’enrichir mutuellement pour prédire, visualiser et comprendre la dynamique de la molécule la plus polyvalente du vivant.
Nature Communications 11November 2025
https://doi.org/10.1038/s41467-025-65502-8