Highly accurate protein structure prediction for the human proteome
Nature 22 July 2021
10.1038/s41586-021-03819-2
Partners use AlphaFold, the AI system recognised last year as a solution to the protein structure prediction problem, to release more than 350,000 protein structure predictions including the entire human proteome to the scientific community.
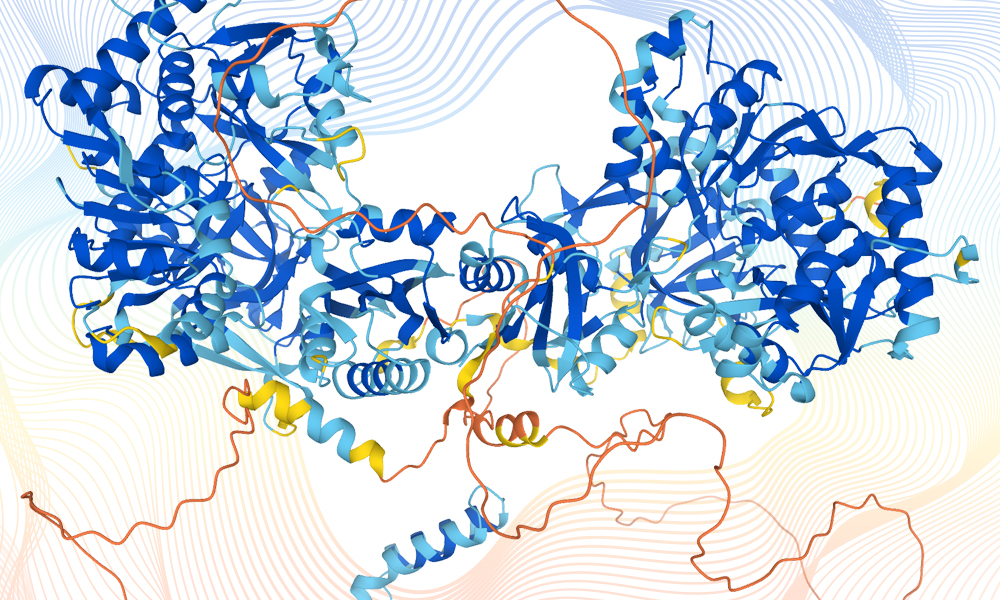
DeepMind today announced its partnership with the European Molecular Biology Laboratory (EMBL), Europe’s flagship laboratory for the life sciences, to make the most complete and accurate database yet of predicted protein structure models for the human proteome. This will cover all ~20,000 proteins expressed by the human genome, and the data will be freely and openly available to the scientific community. The database and artificial intelligence system provide structural biologists with powerful new tools for examining a protein’s three-dimensional structure, and offer a treasure trove of data that could unlock future advances and herald a new era for AI-enabled biology.
AlphaFold’s recognition in December 2020 by the organisers of the Critical Assessment of protein Structure Prediction (CASP) benchmark as a solution to the 50-year-old grand challenge of protein structure prediction was a stunning breakthrough for the field. The AlphaFold Protein Structure Database builds on this innovation and the discoveries of generations of scientists, from the early pioneers of protein imaging and crystallography, to the thousands of prediction specialists and structural biologists who’ve spent years experimenting with proteins since. The database dramatically expands the accumulated knowledge of protein structures, more than doubling the number of high-accuracy human protein structures available to researchers. Advancing the understanding of these building blocks of life, which underpin every biological process in every living thing, will help enable researchers across a huge variety of fields to accelerate their work.
Last week, the methodology behind the latest highly innovative version of AlphaFold, the sophisticated AI system announced last December that powers these structure predictions, and its open source code were published in Nature. Today’s announcement coincides with a second Nature paper that provides the fullest picture of proteins that make up the human proteome, and the release of 20 additional organisms that are important for biological research.
“Our goal at DeepMind has always been to build AI and then use it as a tool to help accelerate the pace of scientific discovery itself, thereby advancing our understanding of the world around us,” said DeepMind Founder and CEO Demis Hassabis, PhD. “We used AlphaFold to generate the most complete and accurate picture of the human proteome. We believe this represents the most significant contribution AI has made to advancing scientific knowledge to date, and is a great illustration of the sorts of benefits AI can bring to society.”
The ability to predict a protein’s shape computationally from its amino acid sequence – rather than determining it experimentally through years of painstaking, laborious and often costly techniques – is already helping scientists to achieve in months what previously took years.
“The AlphaFold database is a perfect example of the virtuous circle of open science,” said EMBL Director General Edith Heard. “AlphaFold was trained using data from public resources built by the scientific community so it makes sense for its predictions to be public. Sharing AlphaFold predictions openly and freely will empower researchers everywhere to gain new insights and drive discovery. I believe that AlphaFold is truly a revolution for the life sciences, just as genomics was several decades ago and I am very proud that EMBL has been able to help DeepMind in enabling open access to this remarkable resource.”
AlphaFold is already being used by partners such as the Drugs for Neglected Diseases Initiative (DNDi), which has advanced their research into life-saving cures for diseases that disproportionately affect the poorer parts of the world, and the Centre for Enzyme Innovation (CEI) is using AlphaFold to help engineer faster enzymes for recycling some of our most polluting single-use plastics. For those scientists who rely on experimental protein structure determination, AlphaFold’s predictions have helped accelerate their research. For example, a team at the University of Colorado Boulder is finding promise in using AlphaFold predictions to study antibiotic resistance, while a group at the University of California San Francisco has used them to increase their understanding of SARS-CoV-2 biology.
The AlphaFold Protein Structure Database builds on many contributions from the international scientific community, as well as AlphaFold’s sophisticated algorithmic innovations and EMBL-EBI’s decades of experience in sharing the world’s biological data. DeepMind and EMBL’s European Bioinformatics Institute (EMBL-EBI) are providing access to AlphaFold’s predictions so that others can use the system as a tool to enable and accelerate research and open up completely new avenues of scientific discovery.
“This will be one of the most important datasets since the mapping of the Human Genome,” said EMBL Deputy Director General, and EMBL-EBI Director Ewan Birney. “Making AlphaFold predictions accessible to the international scientific community opens up so many new research avenues, from neglected diseases to new enzymes for biotechnology and everything in between. This is a great new scientific tool, which complements existing technologies, and will allow us to push the boundaries of our understanding of the world.”
In addition to the human proteome, the database launches with ~350,000 structures including 20 biologically-significant organisms such as E.coli, fruit fly, mouse, zebrafish, malaria parasite and tuberculosis bacteria. Research into these organisms has been the subject of countless research papers and numerous major breakthroughs. These structures will enable researchers across a huge variety of fields – from neuroscience to medicine – to accelerate their work.
The database and system will be periodically updated as we continue to invest in future improvements to AlphaFold, and over the coming months we plan to vastly expand the coverage to almost every sequenced protein known to science – over 100 million structures covering most of the UniProt reference database.
To learn more, please see the Nature papers describing our full method and the human proteome, and read the Authors’ Notes. See the open-source code to AlphaFold if you want to view the workings of the system, and Colab notebook to run individual sequences. To explore the structures, visit EMBL-EBI’s searchable database that is open and free to all.
Jacques Dubochet, Nobel Laureate for Chemistry 2017, former Group Leader at EMBL
“I love to know that the collaboration between DeepMind and EMBL will make all the knowledge about protein structure open to all.”
Paul Nurse, Nobel Laureate for Physiology or Medicine 2001, Director of the Francis Crick Institute and Chair of EMBL Science Advisory Committee
“With this resource freely and openly available, the scientific community will be able to draw on collective knowledge to accelerate discovery, ushering in a new era for AI-enabled biology.”
Venki Ramakrishnan, Nobel Laureate for Chemistry 2009 and former President of the Royal Society
“This computational work represents a stunning advance on the protein-folding problem, a 50-year-old grand challenge in biology.”
Prof. Dame Janet Thornton, Director Emeritus of EMBL-EBI
“This contributes to our knowledge and understanding of living systems, with all the opportunities for humanity this will unlock.”
This post was originally published on EMBL-EBI News
DeepMind gab heute seine Partnerschaft mit dem Europäischen Laboratorium für Molekularbiologie (EMBL), Europas Vorzeigelabor für Biowissenschaften, bekannt, um die bisher vollständigste und genaueste Datenbank der rund 20 000 Proteine und ihrer vorausberechneten Strukturen aus dem menschlichen Genom, also das vollständige menschlichen Proteom, der wissenschaftliche Gemeinschaft frei zur Verfügung zu stellen. Die Datenbank und die Anwendung künstlicher/artifizieller Intelligenz (AI) bieten Strukturbiologen leistungsstarke neue Werkzeuge für die Untersuchung dreidimensionaler Strukturen von Proteinen. Sie eröffnen damit eine Fundgrube an Daten, die künftige Forschung und Erkenntnisse ermöglichen und eine neue Ära der AI-gestützten Biologie einläuten können.
Die Anerkennung von AlphaFold als Lösung für die 50 Jahre alte große Herausforderung der Proteinstrukturvorhersage im Dezember 2020 durch die Organisatoren des CASP-Benchmarks (Critical Assessment of Protein Structure Prediction) stellte einen signifikanter Durchbruch in der Forschung dar. Die AlphaFold-Proteinstrukturdatenbank baut auf dieser Innovation und den Entdeckungen von Generationen von WissenschaftlerInnen auf: von den frühen Pionieren der Proteinforschung und kristallographie bis hin zu den tausenden VorhersagespezialistInnen und StrukturbiologInnen, die seit Jahren Proteinstrukturen erforschen. Durch die Datenbank wird die Anzahl der hochpräzise dargestellten menschlichen Proteinstrukturen, die Forschenden zur Verfügung stehen, mehr als verdoppelt. AlphaFold erweitert damit das gesammelte Wissen über Proteinstrukturen drastisch. Ein besseres Verständnis dieser Bausteine des Lebens, die jedem biologischen Prozess in jedem Lebewesen zugrunde liegen, wird es Forschenden in den verschiedensten Bereichen ermöglichen, ihre Arbeit bedeutend zu beschleunigen.
Letzte Woche wurde die Methodik hinter der neuesten, hochinnovativen Version von AlphaFold, dem hochentwickelten AI-System, das Strukturvorhersagen ermöglicht, und dessen Open-Source-Code in der wissenschaftliche Fachzeitschrift Nature veröffentlicht. Die heutige Ankündigung fällt mit einer zweiten Veröffentlichung in der Nature zusammen, die nicht nur das umfassendste Bild der Proteine im menschlichen Proteom beschreibt, sondern auch die Proteinstrukturen 20 weiterer Organismen, die für die biologische Forschung bedeutend sind.
“Unser Ziel bei DeepMind war es immer, AI zu entwickeln und sie dann als Werkzeug zu nutzen, um das Tempo wissenschaftlicher Entdeckungen zu beschleunigen und damit unser Verständnis der Welt um uns herum zu verbessern”, sagte DeepMind-Gründer und CEO Demis Hassabis, PhD. “Wir haben AlphaFold verwendet, um das vollständigste und genaueste Bild des menschlichen Proteoms zu erstellen. Wir glauben, dass dies der bedeutendste Beitrag ist, den AI bisher zum Fortschritt wissenschaftlicher Erkenntnisse geleistet hat, und es ist ein großartiges Beispiel für die Art von Nutzen, den AI der Gesellschaft bringen kann.”
Die Möglichkeit, die Form eines Proteins nun rechnerisch auf Grund seiner Aminosäuresequenz vorherzusagen – anstatt sie experimentell durch mühsame und oft kostspielige Techniken zu bestimmen – hilft WissenschaftlerInnen bereits jetzt, in wenigen Monaten zu erreichen, was früher Jahre dauerte.
“Die AlphaFold-Datenbank ist ein perfektes Beispiel für den kontinuierlichen Erfolgszyklus von offener Wissenschaft”, sagte EMBL-Generaldirektorin Edith Heard. “AlphaFold wurde mit Datensätzen aus öffentlichen Ressourcen gefüttert, die von der wissenschaftlichen Gemeinschaft bereitgestellt wurden. Es ist also nur konsequent, dass seine Vorhersagen wiederum frei zugänglich gemacht werden. Die offene und freie Weitergabe der AlphaFold-Vorhersagen wird es Forschenden überall auf der Welt ermöglichen, neue Erkenntnisse zu gewinnen und Entdeckungen voranzutreiben. Ich gehe davon aus, dass AlphaFold eine wirkliche Revolution für die Biowissenschaften ist, so wie es die Genomik vor einigen Jahrzehnten war, und ich bin sehr stolz darauf, dass EMBL DeepMind dabei helfen konnte, den offenen Zugang zu dieser bemerkenswerten Ressource zu ermöglichen.”
AlphaFold wird bereits von Partnern wie der Drugs for Neglected Diseases Initiative (DNDi) eingesetzt, die damit ihre Forschung nach lebensrettenden Heilmitteln für Krankheiten vorantreibt, welche unverhältnismäßig stark wirtschaftlich benachteiligte Teilen der Welt betreffen. Das Centre for Enzyme Innovation (CEI) wiederum nutzt AlphaFold, um schnellere Enzyme für das Recycling einiger der umweltschädlichsten Einwegkunststoffe zu entwickeln. Auch WissenschaftlerInnen, die ihre Forschung auf die experimentelle Bestimmung von Proteinstrukturen basieren, helfen die Vorhersagen von AlphaFold bereits dabei, ihre Arbeit zu beschleunigen. So nutzt zum Beispiel ein Forschungsteam an der University of Colorado Boulder die AlphaFold-Vorhersagen, um Antibiotikaresistenzen zu untersuchen, während eine Gruppe an der University of California San Francisco sie verwendet, um ihr Verständnis der Biologie von SARS-CoV-2 zu verbessern.
Die AlphaFold Protein Structure Database baut sowohl auf vielen früheren Beiträgen der internationalen wissenschaftlichen Gemeinschaft auf, als auch auf den hochentwickelten algorithmischen Innovationen von AlphaFold und der jahrzehntelangen Erfahrung des European Bioinformatics Institute (EMBL-EBI) bei der freien Bereitstellung von weltweit gesammelten biologischen Daten. DeepMind und EMBL-EBI stellen den Zugang zu den Vorhersagen von AlphaFold offen zur Verfügung, damit die wissenschaftliche Gemeinschaft das System als Werkzeug dazu nutzen kann, weitere Forschung zu ermöglichen, zu beschleunigen und völlig neue Wege der wissenschaftlichen Entdeckung zu eröffnen.
“AlphaFold wird sich als einer der wichtigsten Datensätze seit der Kartierung des menschlichen Genoms herausstellen”, sagte der stellvertretende EMBL-Generaldirektor und EMBL-EBI-Direktor Ewan Birney. “Die AlphaFold-Vorhersagen der internationalen wissenschaftlichen Gemeinschaft offen zugänglich zu machen, eröffnet so viele neue Forschungsmöglichkeiten. Von vernachlässigten Krankheiten bis hin zu neuen Enzymen für die Biotechnologie und allem, was dazwischen liegt. Dies ist ein großartiges neues wissenschaftliches Werkzeug, das bestehende Technologien ergänzt und es uns ermöglichen wird, die Grenzen unseres Verständnisses der Welt zu erweitern.”
Zusätzlich zum menschlichen Proteom geht die Datenbank mit rund 350.000 weiteren Proteinstrukturen online, darunter Proteine von 20 biologisch bedeutsame Organismen wie der Fruchtfliege, der Maus, des Zebrafisches, des Malariaparasiten sowie von E.coli- und Tuberkulosebakterien. Die Forschung an diesen Organismen war bereits Grundlage zahlreicher bedeutender wissenschaftlicher Durchbrüche und entsprechender Veröffentlichungen. Ihre nun veröffentlichte Proteinstrukturen werden es Forschern in den verschiedensten Bereichen – von den Neurowissenschaften bis zur Medizin – ermöglichen, ihre Forschung schneller voranzutreiben.
Die beteiligten Partner werden in den kommenden Monaten kontinuierlich in die Verbesserung von AlphaFold investieren und die Datenbank und das dahinterliegende System regelmäßig aktualisieren. So soll die Abdeckung auf fast alle sequenzierten Proteine, die der Wissenschaft bekannt sind, erweitert werden – über 100 Millionen Strukturen, die den Großteil der UniProt-Referenzdatenbank abdecken.
Weitere Informationen können Sie dem Fachartikel zum Thema in der Nature entnehmen, der die vollständige Methodik und das menschliche Proteom beschreibt sowie den Anmerkungen der Autoren. Besuchen Sie den Open-Source-Code zu AlphaFold, wenn Sie sich die Funktionsweise des Systems ansehen möchten und Colab notebook, um einzelne Sequenzen zu untersuchen. Um die Strukturen zu erforschen, besuchen Sie die Datenbank von EMBL-EBI, die offen und kostenfrei zugänglich ist.
DeepMind a annoncé aujourd’hui son partenariat avec le Laboratoire européen de biologie moléculaire (EMBL), le laboratoire de référence pour les sciences de la vie en Europe, afin de mettre à disposition de manière libre et gratuite à l’ensemble de la communauté scientifique la base de données la plus complète et la plus précise à ce jour de prédictions de modèles de structures des protéines du protéome humain. Ceci équivaut à un ensemble d’environ 20 000 protéines exprimées par le génome humain. Ces données seront mises à disposition de la communauté scientifique de manière libre et gratuite. La base de données et le système d’intelligence artificielle fournissent aux chercheurs en biologie structurale de nouveaux et puissants outils pour examiner la structure tridimensionnelle d’une protéine, et offrent une véritable mine d’informations qui pourrait débloquer de futures avancées et annoncer une nouvelle ère pour la biologie assistée par l’intelligence artificielle.
La reconnaissance d’AlphaFold en décembre 2020 par les organisateurs de l’Evaluation critique de la prédiction de la structure des protéines (ou CASP, Critical Assessment of protein Structure Prediction, en anglais) comme une solution au grand défi, vieux de 50 ans, de la prédiction de la structure des protéines, a constitué une remarquable avancée dans ce domaine. La base de données sur la structure des protéines AlphaFold s’appuie sur cette innovation et sur les découvertes de générations de scientifiques, depuis les premiers pionniers de l’imagerie et de la cristallographie des protéines, jusqu’aux milliers de spécialistes de la prédiction et de chercheurs en biologie structurale qui ont, depuis lors, passé des années à réaliser des expérimentations sur les protéines. La base de données élargit considérablement les connaissances accumulées sur les structures protéiques, en faisant plus que doubler le nombre de structures protéiques humaines de haute précision mises à disposition des chercheurs. L’amélioration de la compréhension de ces éléments constitutifs de la vie, qui sont à la base de tous les processus biologiques de chaque être vivant, permettra aux chercheurs d’accélérer leurs travaux dans un très grand nombre de domaines.
La semaine dernière, la méthodologie à la base de la dernière version hautement innovante d’AlphaFold – le sophistiqué système d’intelligence artificielle annoncé en décembre dernier qui permet ces prédictions de structure – et son code source ouvert (open source code), ont été publiés dans Nature. L’annonce faite aujourd’hui coïncide avec la publication d’un deuxième article dans Nature qui donne l’image la plus complète des protéines qui composent le protéome humain, et la publication de 20 organismes supplémentaires importants pour la recherche en biologie.
“Notre objectif à DeepMind a toujours été de construire l’intelligence artificielle et de l’utiliser comme un outil pour aider à accélérer le rythme de la découverte scientifique, faisant ainsi progresser notre compréhension du monde qui nous entoure”, a déclaré le fondateur et PDG de DeepMind, Demis Hassabis, PhD. “Nous avons utilisé AlphaFold pour générer l’image la plus complète et la plus précise du protéome humain. Nous pensons que cela représente, à ce jour, la contribution la plus importante de l’intelligence artificielle à l’avancement des connaissances scientifiques, et illustre parfaitement le type d’avantages que celle-ci peut apporter à la société.”
La possibilité de prédire la forme d’une protéine par ordinateur à partir de sa séquence d’acides aminés – plutôt que de la déterminer expérimentalement par des années de techniques minutieuses, ardues et souvent coûteuses – aide déjà les scientifiques à réaliser en quelques mois ce qui prenait auparavant des années.
“La base de données AlphaFold est un exemple parfait du cercle vertueux de la science ouverte”, a déclaré Edith Heard, directrice générale de l’EMBL. “AlphaFold a été formé en utilisant des données provenant de ressources publiques construites par la communauté scientifique, il est donc logique que ces prédictions soient rendues publiques. Le partage ouvert et gratuit des prédictions d’AlphaFold permettra aux chercheurs du monde entier d’acquérir de nouvelles connaissances et de stimuler la découverte. Je pense qu’AlphaFold est véritablement une révolution pour les sciences de la vie, tout comme l’a été la génomique il y a plusieurs décennies, et je suis très fière que l’EMBL ait pu aider DeepMind à rendre publique cette ressource remarquable”.
AlphaFold est déjà utilisé par des partenaires tels que l’initiative Drugs for Neglected Diseases (DNDi), qui a fait progresser la recherche de traitements permettant de sauver des vies pour des maladies qui touchent de manière disproportionnée les régions les plus pauvres du monde, et le Centre for Enzyme Innovation (CEI) qui utilise AlphaFold pour aider à concevoir des enzymes plus rapides pouvant servir à recycler certains des plastiques à usage unique les plus polluants. Pour les scientifiques qui dépendent de la détermination expérimentale de la structure des protéines, les prédictions d’AlphaFold ont permis d’accélérer leurs recherches. Par exemple, une équipe de l’université du Colorado à Boulder trouve prometteuse l’utilisation des prédictions d’AlphaFold pour étudier la résistance aux antibiotiques, tandis qu’un groupe de l’université de Californie à San Francisco les a utilisées pour mieux comprendre la biologie du SARS-CoV-2.
La base de données sur la structure des protéines AlphaFold s’appuie sur de nombreuses contributions de la communauté scientifique internationale, ainsi que sur les innovations algorithmiques sophistiquées d’AlphaFold et sur les décennies d’expérience de l’EMBL-EBI en matière de partage des données biologiques au niveau mondial. DeepMind et l’Institut européen de bioinformatique de l’EMBL (EMBL-EBI) donnent accès aux prédictions d’AlphaFold afin que d’autres puissent utiliser ce système comme un outil permettant et accélérant la recherche, et ouvrant de toutes nouvelles voies à la découverte scientifique.
“Ce sera l’un des ensembles de données les plus importants depuis la cartographie du génome humain”, a déclaré Ewan Birney, directeur général adjoint de l’EMBL et directeur de l’EMBL-EBI. “Rendre les prédictions d’AlphaFold accessibles à la communauté scientifique internationale ouvre énormément de nouvelles voies de recherche, depuis les maladies négligées jusqu’aux nouvelles enzymes pour la biotechnologie, et pour une multitude d’autres domaines. Il s’agit d’un nouvel outil scientifique formidable, qui complète les technologies existantes, et qui nous permettra de repousser les limites de notre compréhension du monde.”
Au-delà du protéome humain, la base de données comprend au total environ 350 000 structures de protéines, dont celles de 20 organismes biologiquement significatifs tels que E.coli, la mouche du vinaigre (drosophile), la souris, le poisson zèbre, le parasite du paludisme et la bactérie de la tuberculose. Les recherches portant sur ces organismes ont fait l’objet d’innombrables articles de recherche et de nombreuses avancées majeures. Ces structures permettront aux chercheurs d’une grande variété de domaines – des neurosciences à la médecine – d’accélérer leurs travaux.
La base de données et le système seront périodiquement mis à jour, au fur et à mesure que nous continuons à investir dans des améliorations futures d’AlphaFold. Au cours des prochains mois, nous prévoyons d’étendre considérablement la couverture à presque toutes les protéines séquencées connues de la science – plus de 100 millions de structures couvrant la majeure partie de la base de données de référence UniProt.
Pour plus de détails, il est possible de consulter les articles de Nature décrivant notre méthode complète et le protéome humain, et de lire les notes des auteurs ici. Il est également possible de consulter le code open source d’AlphaFold pour voir le fonctionnement du système, et le Colab notebook pour exécuter des séquences individuelles. Pour explorer les structures, il est possible de visiter la base de données consultable de l’EMBL-EBI, qui est ouverte et gratuite pour tous.
DeepMind ha annunciato oggi la sua partnership con il Laboratorio Europeo di Biologia Molecolare (EMBL), il principale laboratorio europeo per le scienze della vita, per il lancio del database più completo e accurato per la predizione delle strutture del proteoma umano. Questi dati – comprese le strutture delle circa 20.000 proteine espresse dal genoma umano – saranno disponibili tramite accesso aperto e libero alla comunità scientifica. Il database e il sistema di intelligenza artificiale forniscono ai biologi strutturali nuovi potenti strumenti per esaminare la struttura tridimensionale delle proteine, e offrono dati preziosi che potrebbero accelerare i progressi futuri e annunciare una nuova era per la biologia supportata dall’intelligenza artificiale (IA).
Lo scorso dicembre, Il riconoscimento di AlphaFold da parte degli organizzatori dell’iniziativa “Critical Assessment of protein Structure Prediction (CASP)” come soluzione alla grande sfida che risale a 50 anni fa della predizione della struttura delle proteine è stata una svolta sorprendente nel settore. L’AlphaFold Protein Structure Database si basa su questa innovazione e sulle scoperte di generazioni di scienziati, dai primi pionieri dell’imaging e della cristallografia, alle migliaia di specialisti che hanno passato anni a studiare le proteine. Il database aumenta moltissimo la conoscenza acquisita finora, più che raddoppiando il numero di strutture proteiche umane predette con grande precisione e ora messe a disposizione dei ricercatori di tutto il mondo. Comprendere più in dettaglio come funzionano questi elementi costitutivi della vita, alla base di ogni processo biologico negli esseri viventi, permetterà ai ricercatori di velocizzare il loro lavoro in moltissimi campi.
La scorsa settimana, la metodologia alla base dell’ultima versione altamente innovativa di AlphaFold – il sofisticato sistema di IA annunciato lo scorso dicembre che guida le previsioni di struttura – e il suo codice open source sono stati pubblicati sulla rivista Nature. L’annuncio di oggi coincide con un secondo articolo su Nature che fornisce il quadro più completo delle proteine che compongono il proteoma umano, e la pubblicazione delle predizioni delle strutture proteiche di altri 20 organismi importanti per la ricerca biologica.
“Il nostro obiettivo a DeepMind è sempre stato quello di costruire IA e poi usarla come strumento per contribuire ad accelerare il passo della ricerca scientifica, facendo così progredire la nostra comprensione del mondo che ci circonda”, ha detto il fondatore e CEO di DeepMind Demis Hassabis “Abbiamo usato AlphaFold per generare il quadro più completo e accurato possibile del proteoma umano. Crediamo che questo rappresenti il contributo più significativo che l’IA abbia dato al progresso della conoscenza scientifica fino ad oggi, ed è un grande esempio dei benefici che l’IA può portare alla società”.
La capacità di prevedere la forma di una proteina tramite calcoli computazionali partendo dalla sua sequenza di aminoacidi – piuttosto che determinarla sperimentalmente attraverso anni di tecniche minuziose, laboriose e spesso costose – sta già aiutando gli scienziati a raggiungere in mesi ciò che prima richiedeva anni.
“Il database AlphaFold è un perfetto esempio del circolo virtuoso alimentato dalla scienza aperta”, ha detto la direttrice generale dell’EMBL Edith Heard. “AlphaFold è stato istruito usando dati provenienti da basi di dati pubbliche costruite dalla comunità scientifica, quindi ha senso che anche le sue previsioni siano pubbliche. Condividere le previsioni di AlphaFold apertamente e liberamente permetterà ai ricercatori di tutto il mondo di ottenere nuove informazioni e di avanzare rapidamente nelle loro scoperte. Credo che AlphaFold sia davvero una rivoluzione per le scienze della vita, proprio come lo è stata la genomica diversi decenni fa, e sono molto orgogliosa che l’EMBL sia stato in grado di aiutare DeepMind a garantire l’accesso aperto a questa straordinaria risorsa”.
AlphaFold è già utilizzato da partner come la Drugs for Neglected Diseases Initiative (DNDi), per avanzare la ricerca di cure salvavita per malattie che colpiscono in modo sproporzionato le parti più povere del mondo; inoltre, il Centre for Enzyme Innovation (CEI) sta utilizzando AlphaFold per aiutare a progettare enzimi più veloci per il riciclaggio di alcune delle nostre plastiche monouso più inquinanti. Le previsioni di AlphaFold stanno inoltre contribuendo ad agilizzare la ricerca degli scienziati che si affidano alla determinazione sperimentale della struttura delle proteine. Per esempio, un team dell’Università del Colorado Boulder sta utilizzando le previsioni di AlphaFold per studiare la resistenza agli antibiotici, mentre un gruppo dell’Università della California San Francisco le ha usate per studiare la biologia del SARS-CoV-2.
L’ AlphaFold Protein Structure Database si basa sui numerosi contributi della comunità scientifica internazionale, sulle sofisticate innovazioni degli algoritmi di AlphaFold e su decenni di esperienza di EMBL-EBI nella condivisione mondiale dei dati biologici. DeepMind e l’Istituto Europeo di Bioinformatica dell’EMBL (EMBL-EBI) faciliteranno l’accesso alle previsioni di AlphaFold in modo che tutti possano utilizzare il sistema come strumento per consentire e accelerare la ricerca e aprire strade completamente nuove alla scoperta scientifica.
“Questo sarà uno degli insiemi di dati più importanti dalla mappatura del genoma umano”, ha detto il vice direttore generale dell’EMBL e direttore dell’EMBL-EBI Ewan Birney. “Rendere le predizioni AlphaFold accessibili alla comunità scientifica internazionale apre moltissime nuove strade di ricerca, dalle malattie trascurate ai nuovi enzimi per la biotecnologia e molto altro. Si tratta di un nuovo grande strumento scientifico, che integra le tecnologie esistenti, e ci permetterà di allargare i confini della nostra comprensione del mondo”.
Oltre al proteoma umano, il database viene lanciato con circa 350.000 strutture proteiche tra cui quelle di 20 organismi biologicamente significativi come il batterio E.coli, il moscerino della frutta, il topo, zebrafish, il parassita della malaria e i batteri della tubercolosi. La ricerca su questi organismi è stata oggetto di numerose pubblicazioni scientifiche e scoperte importanti. Queste strutture permetteranno ai ricercatori impegnati in una grande varietà di campi – dalle neuroscienze alla medicina – di accelerare il loro lavoro.
Il database e il sistema saranno aggiornati periodicamente man mano che continueremo a investire nei miglioramenti futuri di AlphaFold, e nei prossimi mesi abbiamo in programma di espandere notevolmente la copertura a quasi tutte le sequenze di proteine note – oltre 100 milioni di strutture che coprono la maggior parte del database di riferimento UniProt.
Per ulteriori dettagli, si prega di consultare gli articoli su Nature che descrivono il nostro metodo completo e il proteoma umano, e leggere le note degli autori qui. Il codice open-source di AlphaFold mostra il funzionamento del sistema e il Colab notebook analizza le singole sequenze. Per esplorare le strutture, visitate il database di ricerca di EMBL-EBI che è aperto e gratuito per tutti.
DeepMind ha anunciado hoy su colaboración con el Laboratorio Europeo de Biología Molecular (EMBL), el principal laboratorio europeo en ciencias de la vida, para proporcionar de manera libre y abierta a la comunidad científica la base de datos de los modelos de predicciones de las estructuras del proteoma humano más completa y precisa hasta la fecha. Esto incluirá alrededor de 20,000 proteínas expresadas por el genoma humano. La base de datos y el sistema de inteligencia artificial brindan a los biólogos estructurales nuevas y poderosas herramientas para examinar la estructura tridimensional de las proteínas, y ofrecen un tesoro de datos que podría abrir el camino a futuros avances y presagiar una nueva era para la biología basada en la inteligencia artificial.
En diciembre de 2020, los organizadores de la evaluación comparativa Critical Assessment of Protein Structure Prediction (CASP) reconocieron AlphaFold como una solución al gran desafío de más de 50 años de predecir la estructura de proteínas, lo que significó un logro asombroso en el campo. La base de datos de estructura de proteínas AlphaFold (AlphaFold Protein Structure Database) se basa en esta innovación y en los descubrimientos de generaciones de científicos y científicas, desde los pioneros y las pioneras de la cristalografía y el análisis de estructura de las proteínas , hasta los miles de especialistas en predicción y biólogos y biólogas estructurales que han pasado años experimentando con proteínas desde entonces y que han compartido sus resultados de forma abierta. La base de datos explota y amplía drásticamente el conocimiento acumulado sobre las estructuras de proteínas, más que duplicando el número de estructuras de proteínas humanas con predicciones de alta precisión disponibles para los investigadores. Avanzar en la comprensión de estos componentes básicos de la vida, que sustentan los procesos biológicos en todos los seres vivos, permitirá a los investigadores de una gran variedad de campos acelerar su trabajo.
La semana pasada se publicó en la revista Nature la metodología de la última e innovadora versión de AlphaFold, el sofisticado sistema de inteligencia artificial anunciado en diciembre pasado que impulsa estas predicciones de estructura, y su código fuente abierto. El anuncio de hoy coincide con un segundo artículo de Nature que proporciona la imagen más completa de las proteínas que componen el proteoma humano, y la publicación de las proteínas de 20 organismos adicionales que son importantes para la investigación biológica.
“Nuestro objetivo en DeepMind siempre ha sido construir inteligencia artificial y utilizarla como una herramienta para ayudar a acelerar el ritmo del descubrimiento científico, y mejorar así el conocimiento del mundo que nos rodea”, dijo el fundador y director ejecutivo de DeepMind, el Dr. Demis Hassabis. “Hemos utilizado AlphaFold para generar la imagen más completa y precisa del proteoma humano. Creemos que esta es la contribución más significativa que ha hecho la inteligencia artificial al avance del conocimiento científico hasta la fecha, y es un gran ejemplo de los tipos de beneficios que la inteligencia artificial puede aportar a la sociedad”.
La capacidad de predecir computacionalmente la forma de una proteína a partir de su secuencia de aminoácidos, en lugar de tener que determinarla experimentalmente con técnicas minuciosas, laboriosas, y a menudo costosas, ya está ayudando a los científicos a lograr en meses lo que antes requería años de trabajo.
“La base de datos AlphaFold es un ejemplo perfecto del círculo virtuoso de la ciencia abierta”, dijo la directora general del EMBL, Edith Heard. “AlphaFold ha sido entrenado utilizando datos de recursos públicos creados por la comunidad científica, por lo que tiene sentido que sus predicciones sean públicas. Compartir las predicciones de AlphaFold de forma abierta y gratuita permitirá a los investigadores de todo el mundo obtener nuevos conocimientos e impulsar nuevos descubrimientos. Creo que AlphaFold es una verdadera revolución para las ciencias de la vida, así como fue la genómica hace varias décadas y estoy muy orgullosa de que el EMBL haya podido ayudar a DeepMind a permitir el acceso abierto a este recurso extraordinario”.
AlphaFold ya está siendo utilizado por socios como la Iniciativa de Medicamentos para Enfermedades Desatendidas (DNDi, por sus siglas en inglés), que ha avanzado en su investigación sobre curas que salvan vidas para enfermedades que afectan de manera desproporcionada a las zonas más pobres del mundo, o el Centro de Innovación Enzimática (CEI) que utiliza AlphaFold para ayudar a diseñar enzimas más rápidas para reciclar algunos de los plásticos más contaminantes de un solo uso. AlphaFold ha ayudado a acelerar la investigación de aquellos científicos y científicas que trabajan en la determinación experimental de la estructura de las proteínas. Por ejemplo, un equipo de la Universidad de Colorado en Boulder utiliza las predicciones de AlphaFold para estudiar la resistencia a los antibióticos, mientras que un grupo de la Universidad de California en San Francisco las ha utilizado para estudiar la biología del SARS-CoV-2.
La base de datos de estructura de proteínas AlphaFold está basada en muchas contribuciones de la comunidad científica internacional, así como en las refinadas innovaciones algorítmicas de AlphaFold y en las décadas de experiencia del Instituto Europeo de Bioinformática del EMBL (EMBL-EBI) compartiendo datos biológicos mundiales. DeepMind y el EMBL-EBI están dando libre acceso a las predicciones de AlphaFold para que cualquiera pueda usar el sistema con el fin de permitir y acelerar la investigación y explorar nuevas vías de conocimiento científico.
“Este será uno de los conjuntos de datos más importantes desde el mapa del Genoma Humano”, ha dicho el Director General Adjunto del EMBL y el director del EMBL-EBI, Ewan Birney. “Hacer que las predicciones de AlphaFold sean accesibles a la comunidad científica internacional abre muchas nuevas vías de investigación, desde enfermedades desatendidas hasta nuevas enzimas para la biotecnología y mucho más. Esta es una nueva y gran herramienta científica, que complementa las tecnologías existentes y nos permitirá ampliar los límites de nuestra comprensión del mundo “.
Entre las primeras más de 350.000 estructuras publicadas en la base de datos, además del proteoma humano, están las proteínas de 20 organismos biológicamente significativos como E. coli, la mosca de la fruta, el ratón, el pez cebra, el parásito de la malaria y las bacterias de la tuberculosis. Se han realizado muchas investigaciones importantes sobre estos organismos, y tener estas estructuras a disposición permitirá a muchos investigadores de campos muy diferentes, desde la neurociencia hasta la medicina, acelerar su trabajo.
La base de datos y el sistema serán actualizados periódicamente a medida que se continúe invirtiendo en mejoras futuras de AlphaFold, y en los próximos meses se planea expandir enormemente la cobertura a casi todas las proteínas secuenciadas conocidas por la ciencia: más de 100 millones de estructuras que incluyen la mayoría de UniProt, la base de datos referencia.
Para más detalles, se pueden consultar los artículos de Nature que describen el método completo y el proteoma humano, y leer las notas de los autores aquí. Para ver el funcionamiento del sistema, se puede ver el código de fuente abierta para AlphaFold, y el cuaderno Colab para ejecutar secuencias individuales. Para explorar las estructuras, se puede visitar la base de datos de búsqueda del EMBL-EBI, abierta y gratuita para todo el mundo.
Nature 22 July 2021
10.1038/s41586-021-03819-2
Nature 15 July 2021
10.1038/s41586-021-03819-2