Read the latest Issue
Replication cycle of SARS-CoV-2 in 3D
Learning how SARS-CoV-2 hijacks host cell machineries will help in developing therapeutic strategies
As the global coronavirus pandemic continues, scientists are not only trying to find vaccines and drugs to combat it, but also to continuously learn more about the virus itself. “By now we can expect the coronavirus to become seasonal,” explains Ralf Bartenschlager, professor in the Department of Infectious Diseases, Molecular Virology, at Heidelberg University. “Thus, there is an urgent need to develop and implement both prophylactic and therapeutic strategies against this virus.” In a new study, Bartenschlager, assisted by the Schwab team at EMBL Heidelberg and using EMBL’s Electron Microscopy Core Facility, performed a detailed imaging analysis to determine how the virus reprograms infected cells.
Cells that become infected by SARS-CoV-2 die rather quickly, within only 24 to 48 hours. This indicates that the virus harms the human cell in such a way that it is rewired and essentially forced to produce viral progeny. The main aim of the project was therefore to identify the morphological changes within a cell that are inherent to this reprogramming. “To develop drugs which suppress the viral replication and thereby the consequence of the infection, as well as the virus-induced cell death, is key to have a better understanding of the biological mechanisms driving the virus’ replication cycle,” explains Bartenschlager. The team used the imaging facilities at EMBL and state-of-the art imaging techniques to determine the 3D architecture of SARS-CoV-2-infected cells, as well as alterations of cellular architecture caused by the virus.
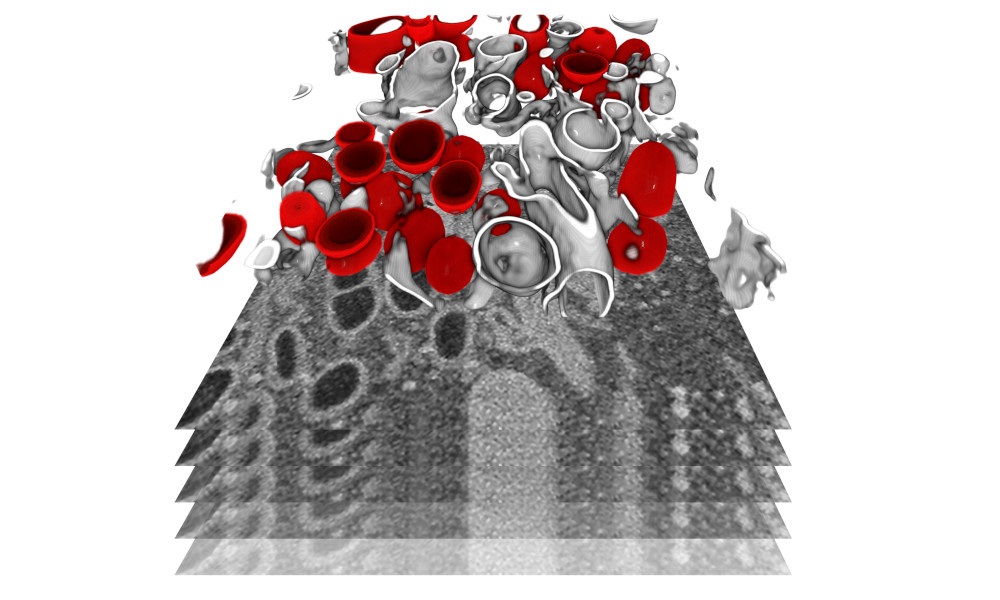
The team was able to create 3D reconstructions of whole cells and their subcellular compartments. “We are providing critical insights into virus-induced structural changes in the studied human cells,” explains Ralf Bartenschlager. The images revealed an obvious and massive change in the endomembrane systems of the infected cells – a system that enables the cell to define different compartments and sites. The virus induces membrane changes in such a way that it can produce its own replication organelles. These are mini replication compartments where the viral genome is amplified enormously. To do this, the virus requires membrane surfaces. These are created by exploiting a cellular membrane system and creating an organelle, which has a very distinct appearance. The scientists describe it as a massive accumulation of bubbles: two membrane layers forming a big balloon. Within these balloons – which form a very shielded compartment – the viral genomes are multiplied and released to become incorporated into new virus particles.
This striking change can be seen in the cells only a few hours after infection. “We saw how and where the virus replicates within the cell, and how it hijacks its host machinery to be released after multiplication,” Schwab says. Until now, little was known about the origin and development of the effects that SARS-CoV-2 causes in the human body. This includes a lack of knowledge about the mechanism by which the infection leads to the death of infected cells. Having this information now will foster the development of therapies reducing virus replication and, thus, disease severity.
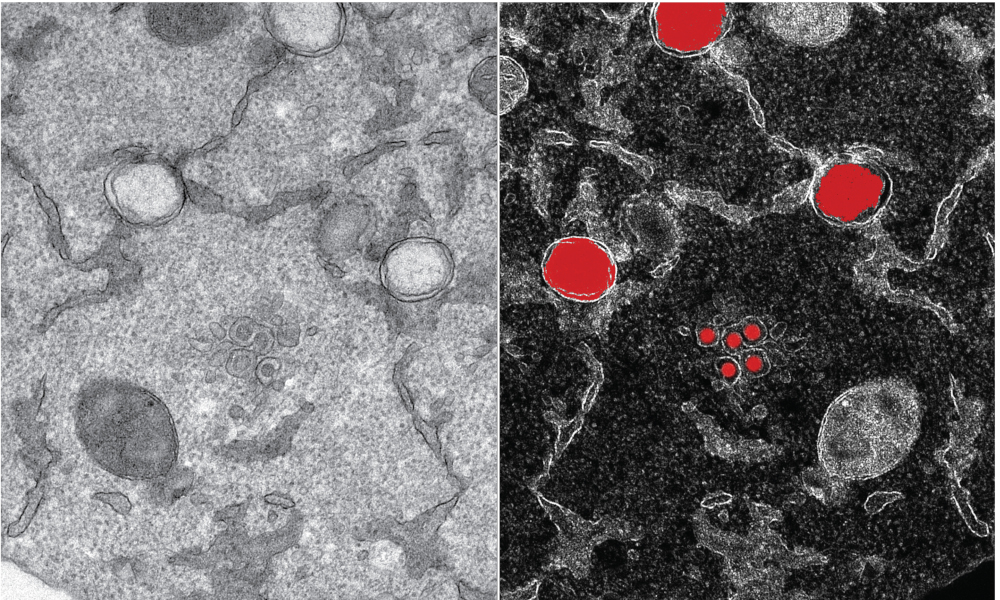
The team has made sure that the collected information and, in particular, the unprecedented repository of 3D structural information about virus-induced substructures can be used by everyone. “I believe we are setting a precedent on the fact that we are sharing all data that we produced with the scientific community. It represents an impressive resource to the community,” says Yannick Schwab. “This way we can support the global effort to study how SARS-CoV-2 interacts with its host.” The team hopes that their collected information will help in the development of antiviral drugs.
The team managed to produce the study in an incredibly short time, despite the challenging circumstances. “Half the world – and of course Heidelberg as well – were in full lockdown and we had to improvise almost on a daily basis to adapt to the situation. Whether at EMBL or from home, all were deeply involved and generously gave their time and deep knowledge,” says Schwab. “The speed at which we worked, and the amount of data that was produced, is remarkable.”
Replikationszyklus von SARS-COV-2 in 3D
Untersuchung wie SARS-CoV-2 die Maschinerien der Wirtszellen übernimmt wird dabei helfen therapeutische Strategien zu entwickeln
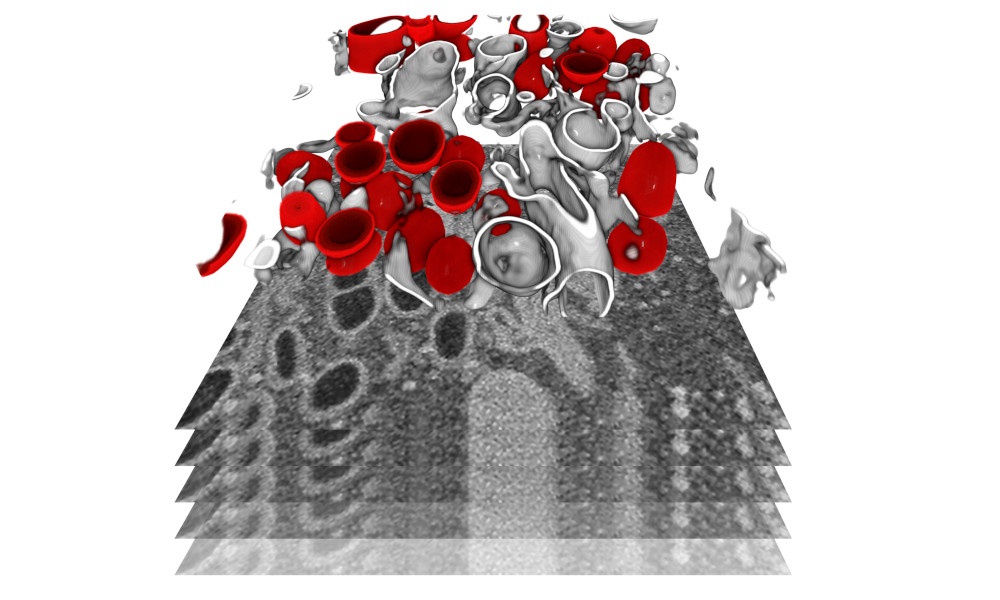
Im Zuge der weltweiten Corona-Pandemie versuchen Wissenschaftler nicht nur Impfstoffe und Medikamente gegen die Krankheit zu finden, sondern auch kontinuierlich mehr über das Virus selbst zu erfahren. „Inzwischen können wir damit rechnen, dass das Coronavirus saisonal wird,“ erklärt Ralf Bartenschlager, Professor in der Abteilung für Infektionskrankheiten der Universität Heidelberg. „Es ist daher dringend notwendig, sowohl prophylaktische als auch therapeutische Strategien gegen dieses Virus zu entwickeln und umzusetzen.“ In einer neuen Studie führte Bartenschlager daher mit Unterstützung des Schwab-Teams am Europäischen Laboratorium für Molekularbiologie in Heidelberg und unter Verwendung der Elektronenmikroskopie-Anlage des EMBL eine detaillierte bildgebende Analyse durch, um festzustellen, wie das Virus infizierte Zellen umprogrammiert.
Zellen, die durch das Virus infiziert werden, sterben innerhalb von nur 24 bis 48 Stunden ab. Dies deutet darauf hin, dass das Virus die menschliche Zelle in einer Weise schädigt, dass diese umprogrammiert und gezwungen wird, Virenpartikel zu produzieren. Die Identifizierung dieser morphologischen Veränderungen innerhalb einer Zelle war daher das Hauptziel des Projekts. „Um Medikamente zu entwickeln, welche die virale Replikation unterdrücken und damit auch die Folgen der Infektion und des virusinduzierten Zelltods, ist es entscheidend, die biologischen Mechanismen, die den Replikationszyklus des Virus antreiben, besser zu verstehen,“ erklärt Bartenschlager. Deshalb nutzte das Team die bildgebenden Einrichtungen des EMBL und modernste bildgebende Verfahren, um die 3D-Architektur von SARS-CoV-2 sowie die virusbedingten Veränderungen der infizierten Zellen zu bestimmen.
Das Team war in der Lage, 3D-Rekonstruktionen von ganzen Zellen und den subzellulären Kompartimenten zu erstellen. „Wir liefern kritische Einblicke in virusinduzierte Strukturveränderungen in den untersuchten menschlichen Zellen,“ erklärt Bartenschlager. Die Bilder zeigten eine offensichtliche und massive Veränderung in den Endomembransystemen der infizierten Zellen – ein System, das der Zelle dazu dient, verschiedene Abteilungen und Orte zu definieren. Das Virus verändert Membranen auf eine Art und Weise, dass es seine eigenen Replikationsorganellen produzieren kann. Dies sind Mini-Replikationsgefäße, in denen das virale Genom enorm vervielfältigt wird. Dazu benötigt das Virus die Oberfläche von Membranen, und das Virus holt sich diese Membranen von den Zellen. Es baut sie so um, dass eine Organelle entsteht, die ein sehr charakteristisches Aussehen hat. Die Wissenschaftler beschreiben es als eine massive Ansammlung von Blasen: zwei Membranschichten, die einen großen Ballon bilden. Innerhalb dieser Ballons – die ein sehr abgeschirmtes Behältnis bilden – werden die viralen Genome repliziert und vermehrt.
Diese auffälligen Veränderungen zeigen sich bereits wenige Stunden nach der Infektion in den infizierten Zellen. „Wir sehen, wie und wo sich das Virus innerhalb der Zelle vermehrt und wie es seine Wirtszellen manipuliert, um nach der Vermehrung freigesetzt zu werden,“ sagte Yannick Schwab. Über die Entstehung und Entwicklung der Auswirkungen, die das Coronavirus im menschlichen Körper hervorruft, war bisher wenig bekannt. Dazu gehören fehlende Kenntnisse über den Mechanismus, wie die Infektion zum Absterben der infizierten Zellen führt. Mit den jetzt vorhandenen Informationen kann die Entwicklung von Therapien, die die Schwere der Krankheit verringern, beschleunigt werden.

Das Team sorgte dafür, dass die gesammelten Informationen und insbesondere die beispiellose Sammlung von 3D-Strukturinformationen von virusinduzierten Substrukturen von jedermann genutzt werden können. „Ich glaube, dass wir damit einen Präzedenzfall dafür schaffen, dass wir alle Daten, die wir produziert haben, mit der wissenschaftlichen Gemeinschaft teilen. Sie stellen eine beeindruckende Ressource für Wissenschaftler dar,“ betont Yannick Schwab. “Auf diese Weise können wir die weltweiten Bemühungen unterstützen, zu untersuchen, wie SARS-CoV-2 mit seinem Wirt interagiert. Das Team hofft, dass die gesammelten Informationen zur Entwicklung antiviraler Medikamente beitragen werden.
Dem Team ist es trotz der schwierigen Umstände gelungen, die Studie in unglaublich kurzer Zeit zu erstellen. „Die halbe Welt – und natürlich auch Heidelberg – war im Lockdown, und wir mussten fast täglich improvisieren, um uns an die Situation anzupassen. Ob am EMBL oder von zu Hause aus, alle waren extrem motiviert und haben großzügig ihre Zeit und ihr Expertenwissen zur Verfügung gestellt,“ betont Schwab stolz. „Die Geschwindigkeit, mit der wir arbeiteten, und die Menge an Daten, die produziert wurde, ist bemerkenswert.“