Read the latest Issue
Innovative method delivers new insights into the stem cell microenvironment
Innovative method delivers new insights into the stem cell microenvironment
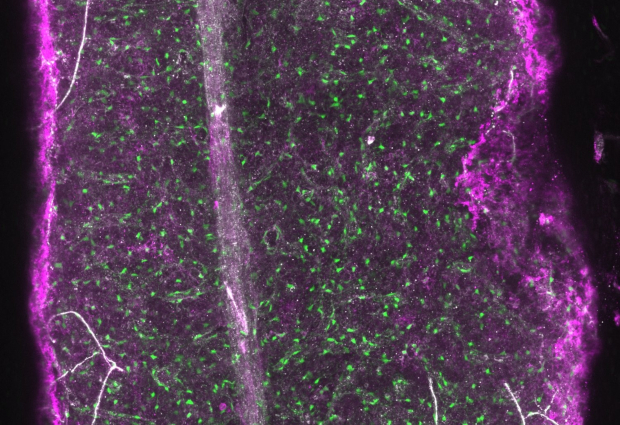
Researchers from EMBL and the German Cancer Research Center (DKFZ) in Heidelberg have developed new methods to reveal the three-dimensional organisation of bone marrow at a single cell level. Since bone marrow harbours blood stem cells responsible for life-long blood generation, these results and the new method provides a novel scientific basis to study blood cancer. The results have been published in Nature Cell Biology.
In the published study the group, led by researchers from EMBL and the DKFZ, present their new methods that permit them to characterise complex organs. The team focused their research on the bone marrow of mice, as it harbours blood stem cells that are responsible for life-long blood production. Because of the ability to influence stem cells and to sustain blood production, there is a growing interest in exploiting the bone marrow environment as a target for novel leukaemia treatments.
“So far, very little was known about how different cells are spatially organised within the bone marrow and how they interact with each other,” explains Chiara Baccin, a post-doc in the Steinmetz Group at EMBL Heidelberg. The newly used methods permitted the team to uncover the cellular composition, the three-dimensional organisation and how the cells interact with each other. In the process the researchers have identified previously unknown cell types, so called “niche cells”, that are of great importance for the regulation of blood stem cells.
In order to understand which cells can be found in the bone marrow, where they are localised and how they might impact on stem cells, the researchers combined single-cell and spatial transcriptomics with novel computational methods. By analysing the RNA content of individual bone marrow cells, the team identified 32 different cell types, including extremely rare and previously unknown cell types. “We believe that these rare ‘niche cells’ establish unique environments in the bone marrow that are required for stem cell function and production of new blood and immune cells,” explains Simon Haas, group leader at the DKFZ and HI-STEM.
Using novel computational methods, the researchers were not only able to determine the organisation of the different cell types in the bone marrow in 3D, but could also predict how cells interact with each other. “It’s the first evidence that spatial interactions in a tissue can be deduced computationally on the basis of genomic data,” explains Lars Velten, staff scientist in the Steinmetz Group.
“Our dataset is publicly accessible to any laboratory in the world and it could be instrumental in refining in vivo studies,” says Lars Steinmetz, group leader at EMBL Heidelberg. The data, which is now already used by different teams all over the world, is accessible in a 3D-atlas via a user-friendly web app. All gathered information is available for free and will in the future facilitate the refinement of genetic studies.
The developed method can be used in different tissues for the study of their 3D organisation. “Our approach is widely applicable and can be used to study the complex pathology of human diseases. We are actually currently in the process of using these approaches on haematological diseases, such as leukaemia,” highlights Andreas Trumpp, managing director of HI-STEM and division head at DKFZ.
Innovative Methode liefert neue Erkenntnisse über die Mikroumgebung der Stammzellen – Knochenmark in 3D
Forscher des Europäischen Laboratoriums für Molekularbiologie (EMBL) und des Deutschen Krebsforschungszentrums (DKFZ) in Heidelberg haben neue Methoden entwickelt, um die dreidimensionale Organisation des Knochenmarks auf Einzelzellebene aufzuzeigen. Da das Knochenmark Blutstammzellen beherbergt, die für die lebenslange Blutbildung verantwortlich sind, bieten diese Ergebnisse und die neue Methode eine neue wissenschaftliche Grundlage für die Untersuchung von Blutkrebs. Die Ergebnisse wurden in Nature Cell Biology veröffentlicht.
In der veröffentlichten Studie stellt die Gruppe um Forscher des EMBL und des DKFZ ihre neuen Methoden vor, mit denen sie komplexe Organe charakterisieren können. Im Mittelpunkt ihrer Forschung steht das Knochenmark von Mäusen, da es Blutstammzellen enthält, die für die lebenslange Blutbildung verantwortlich sind. Aufgrund der Fähigkeit, Stammzellen zu beeinflussen und die Blutproduktion aufrechtzuerhalten, wächst aktuell das Interesse daran, die Umgebung des Knochenmarks als Ziel für neuartige Leukämie-Behandlungen zu nutzen.
“Bislang wusste man sehr wenig darüber, wie verschiedene Zellen im Knochenmark räumlich organisiert sind und wie sie miteinander interagieren”, erklärt Chiara Baccin, Postdoc der Steinmetz-Gruppe am EMBL Heidelberg. Mit den neu eingesetzten Methoden konnte das Team den zellulären Aufbau, die dreidimensionale Organisation und das Zusammenspiel der Zellen aufdecken. Dabei haben die Forscher bisher unbekannte Zelltypen, so genannte “Nischenzellen”, identifiziert, die für die Regulation von Blutstammzellen von großer Bedeutung sind.
Um zu verstehen, welche Zellen im Knochenmark zu finden sind, wo sie lokalisiert sind und wie sie sich auf die Stammzellen auswirken könnten, kombinierten die Forscher die Einzelzell- und die räumliche Transkriptomik mit neuartigen Rechenverfahren. Durch die Analyse des RNA-Gehalts einzelner Knochenmarkszellen identifizierte das Team 32 verschiedene Zelltypen, darunter extrem seltene und bisher unbekannte Zelltypen. “Wir glauben, dass diese seltenen ‘Nischenzellen’ im Knochenmark einzigartige Umgebungen schaffen, die für die Funktion der Stammzellen und die Produktion neuer Blut- und Immunzellen notwendig sind”, erklärt Simon Haas, Gruppenleiter am DKFZ und HI-STEM.
Mit neuartigen Rechenverfahren konnten die Forscher nicht nur die Organisation der verschiedenen Zelltypen im Knochenmark in 3D bestimmen, sondern auch vorhersagen, wie die Zellen miteinander interagieren. “Das ist der erste Nachweis, dass sich räumliche Interaktionen in einem Gewebe rechnerisch aus genomischen Daten ableiten lassen”, erklärt Lars Velten, wissenschaftlicher Mitarbeiter der Steinmetz-Gruppe.
“Unser Datensatz ist für jedes Labor weltweit öffentlich zugänglich und könnte zur Verfeinerung von in-vivo-Studien beitragen”, sagt Lars Steinmetz, Gruppenleiter am EMBL Heidelberg. Die Daten, die nun bereits von verschiedenen Teams auf der ganzen Welt genutzt werden, sind in einem 3D-Atlas über eine benutzerfreundliche Web-Applikation zugänglich. Alle gesammelten Informationen stehen kostenlos zur Verfügung und werden in Zukunft die Verfeinerung der genetischen Studien erleichtern.
Die entwickelte Methode kann in verschiedenen Geweben zur Untersuchung ihrer 3D-Organisation eingesetzt werden. “Unser Ansatz ist breit anwendbar und kann zur Untersuchung der komplexen Pathologie menschlicher Erkrankungen eingesetzt werden. Derzeit sind wir dabei, diese Ansätze auf hämatologische Erkrankungen wie Leukämie anzuwenden”, betont Andreas Trumpp, Geschäftsführer des HI-STEM und Bereichsleiter am DKFZ.