Teil 5: Strukturanalyse
Übersicht
Herzlichen Glückwunsch zum Erreichen des letzten Teils unseres Bioinformatik-Abenteuers! Dank deiner Hilfe wissen wir jetzt, dass das Rhodopsin von einer neuen Art stammt, die eng mit Sardina pilchardus verwandt ist; einem Fisch, der an der europäischen Küste lebt. Die TREC-Forscher*innen sind von deinen Fähigkeiten sehr beeindruckt und haben eine letzte Bitte. Im Rahmen ihrer Forschung möchten sie mehr über die Struktur dieses Rhodopsins erfahren.
In diesem Teil der Aktivität werden wir die strukturellen Aspekte von Proteinen anhand einer Proteindatenbank untersuchen. Die Rhodopsinstruktur dieses neu entdeckten Rhodopsins ist noch nicht verfügbar. Wir können jedoch die Struktur des eng verwandten Rhodopsins von Sardina pilchardus untersuchen, das eine sehr ähnliche Struktur aufweisen sollte. Wir werden uns auf das Rhodopsin der Sardina konzentrieren und die UniProt-Datenbank nutzen, um die Sekundärstruktur dieses Proteins zu untersuchen, insbesondere dessen Alpha-Helices und Beta-Faltblätter. Darüber hinaus werden wir die dreidimensionale Struktur des Sardina-Rhodopsins mit Hilfe der in UniProt integrierten AlphaFold Datenbank untersuchen. Dies wird es uns ermöglichen, vernünftige Schlussfolgerungen über die Struktur des neuen Rhodopsins zu treffen.
Deine Aufgabe
Bitte befolge die unten aufgeführten Schritte:
1. Suche nach dem Sardina Rhodopsin in der UniProt-Datenbank, um die Sekundärstruktur des Proteins zu finden.
2. Untersuche die dreidimensionale Struktur des Sardina Rhodopsins, die von AlphaFold erstellt und in der UniProt-Datenbank angezeigt wird.
3. Versuche, die Fragen auf der Registerkarte “Fragen” zu beantworten.
UniProt and AlphaFold
1. Greife auf die UniProt-Datenbank im untenstehenden Fenster zu.
2. Suchen nach dem Sardina Rhodopsin mit Hilfe des UniProt-Namens (OPSD_SARPI) oder der Hinterlegungsnummer (Q9YGZ0).
3. Auf der Ergebnisseite findest du eine Zusammenfassung aller wissenschaftlichen Informationen, die über das Sardina Rhodopsin verfügbar sind. Untersuche die in der UniProt-Datenbank bereitgestellten Informationen und versuche, einige der Fragen zu beantworten.
4. Scrolle im Fenster der UniProt-Datenbank nach unten zu “Struktur”. Wenn die Struktur nicht angezeigt wird, klicke auf “Click on load the structure viewer”. Du siehst nun eine dreidimensionale Struktur des Sardina Rhodopsins, auch bekannt als Protein-Tertiärstruktur, die von AlphaFold erstellt wurde. Du kannst deinen Mauszeiger benutzen, um die Struktur zu drehen. Außerdem kannst du auf einzelne Aminosäuren innerhalb der Struktur klicken, um weitere Informationen zu erhalten.
5. Schaue dir die dreidimensionale Struktur an und versuche, einige der Fragen zu beantworten.
Fragen
1. Kannst du auf der Grundlage der Informationen aus der UniProt-Datenbank die folgenden Fragen beantworten?
● Wie viele Transmembrandomänen hat das Rhodopsin der Sardina pilchardus und wie sind sie über das Protein verteilt? Diese Informationen finden Sie im Abschnitt “Subcellular Location”.
● Kannst du ausgehend von der vorhergehenden Antwort auf die größere Gruppe der evolutionär verwandten Proteine schließen, zu der unser Rhodopsin gehört?
● Was sind Beispiele für posttranslationale Modifikationen (PTM) des Rhodopsins von Sardina pilchardus? Diese Informationen findest du im Abschnitt “PTM/Processing”.
2. Kannst du auf der Grundlage der von AlphaFold vorhergesagten dreidimensionalen Struktur einige der folgenden Fragen beantworten?
● Welche Form hat das Rhodopsin von Sardina pilchardus im Allgemeinen?
● Versuche, die einzelnen Alpha-Helices und Beta-Faltblätter in der Struktur zu identifizieren. Die Beta-Faltblätter sind wichtig für die Bindung von Retinal. Wo genau befinden sich diese beiden Beta-Faltblätter?
Zusammenfassung
Dank deiner Hilfe konnte das TREC-Team die Funktion und die evolutionären Ursprünge des unbekannten Proteins ermitteln. Außerdem deuten deine Ergebnisse darauf hin, dass dieses Rhodopsin zu einer neuen Art gehören könnte, die eng mit Sardina pilchardus verwandt ist und an der europäischen Küste lebt.
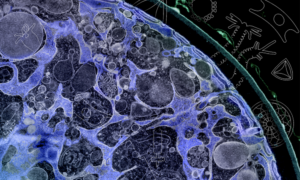
Es sind einige Monate vergangen, seit du dem TREC-Team geholfen hast, und jetzt hat das Team dir überraschenderweise eine weitere E-Mail geschickt! Darin erklären dir die Forscher*innen, dass sie ihre Forschung an diesem neuen Rhodopsin fortgesetzt haben und mit Hilfe des Imaging Centre am EMBL die Proteinstruktur des Rhodopsins dieser neuen Art identifizieren konnten. Dabei verwendeten sie eine Methode namens Kryoelektronenmikroskopie (Kryo-EM) und möchten, dass du der erste bist, der die Rhodopsinstruktur sieht. Hier ist sie:
Ein Antwortblatt mit Beispielantworten für alle Fragen in der Ressource ist auf Anfrage erhältlich. Bitte kontaktieren Sie uns unter schoolvisits@embl.de und geben Sie in Ihrer E-Mail an, dass Sie das Lösungsblatt für diese bestimmte Lernressource benötigen.
Wenn du mehr über Bioinformatik erfahren möchtest, sieh dir diese umfassenden Online-Ressourcen von EMBL-EBI an: https://www.ebi.ac.uk/training/online/courses/introductory-bioinformatics-pathway/.
Aktivitäten-Navigation
● Teil 1: Von der DNA zur Proteinsequenz
● Teil 2: Identität und Funktion von Proteinen
● Teil 3: Multiples Sequenzalignment
Share:
 English
English